Read this post in English here
Hace algunos meses, un colega en Cuba me contactó con unas dudas sobre cómo hacer mapas de riqueza de especies para algunas plantas caribeñas. Yo había escrito una guía breve para estos cálculos, pero solo a partir de puntos de ocurrencia o de mapas de distribución vectoriales. En su caso, había que re-clasificar y sumar varios modelos de MaxEnt para generar una capa de riqueza de especies.
Estuve buscando recursos para recomendarles, pero no encontré nada reciente ni en castellano, así que me puse a armar esta guía. Llevo años sin correr modelos de nicho ecológico (ENM), pero hasta donde sé el proceso sigue siendo el mismo para calcular riqueza.
-
Correr el modelo de distribución potencial (repetir para cada especie)
-
Elegir un umbral y re-clasificar la capa de idoneidad/probabilidad de ocurrencia de cada especie. De valores continuos a valores binarios de presencia/ausencia.
-
Sumar los valores de las capas re-clasificadas para contar el total de especies por pixel.
Desconozco cuál es la práctica actual para hacer ésto, supongo que con QGIS, o con algún programa de ESRI, o directamente con los programas que generan los modelos. Igual aquí propongo una forma bastante sencilla de hacer todo en R.
Con los útimos avances en los métodos espaciales con R, podemos usar stars, sf, y métodos del tidyverse para todos nuestros cálculos y visualizaciones de raster.
Probando el método
Antes que nada, una demostración con datos simulados muy simples. Para ésto hay que generar unos raster de prueba con valores de idoneidad aleatorios.
Esta función crea una matriz de 4x5 con valores aleatorios que luego se convierte en objeto stars, con un nombre asignado por la función adjective_animal del paquete ids.
# cargar paquetes
library(stars) # CRAN v0.5-1
library(sf) # CRAN v0.9-7
library(fs) # CRAN v1.5.0
library(rnaturalearth) # CRAN v0.1.0
library(dplyr) # CRAN v1.0.4
library(purrr) # CRAN v0.3.4
library(ggplot2) # CRAN v3.3.3
library(ids) # CRAN v1.0.1
library(tidyr) # CRAN v1.1.2
library(scico) # CRAN v1.2.0
library(patchwork) # CRAN v1.1.1
library(ggthemes) # CRAN v4.2.0
library(colormap) # CRAN v0.1.4
library(extrafont) # CRAN v0.17
library(ggfx) # [github::thomasp85/ggfx] v0.0.0.9000
# para mayor reproducibilidad
set.seed(20)
# matrix a stars
make_toy_raster <- function(m) {
m <- matrix(sample(seq(0, 100, 1), 20, replace = TRUE), nrow = 5, ncol = 4)
dim(m) <- c(x = 5, y = 4) # named dim
s <- st_as_stars(m)
names(s) <- ids::adjective_animal(1)
s
}Ejecutamos la función 8 veces con purrr::rerun, luego apilamos los objetos con c(), y los combinamos con merge().
# 8 rasters de prueba
toy_data <- rerun(8, make_toy_raster(m))
allsps <- reduce(toy_data, c)
allspsmg <- merge(allsps)Los objetos stars tienen dos elementos muy importantes con los que vamos a estar trabajando: atributos y dimensiones, que son los valores de los pixeles, y los metadatos de las dimensiones que hay en cada capa, respectivamente. Para trabajar con más orden, se le pueden poner nombres a estos elementos, con setNames para los atributos y st_set_dimensions para las coordenadas xy y las especies.
# mejores nombres
allspsmg <- setNames(allspsmg, "prob") %>% st_set_dimensions(names = c("x", "y", "sp"))Así se ve nuestro objeto final:
> allspsmg
stars object with 3 dimensions and 1 attribute
attribute(s):
prob
Min. : 1.00
1st Qu.: 24.00
Median : 51.00
Mean : 50.11
3rd Qu.: 74.00
Max. :100.00
dimension(s):
from to offset delta refsys point
x 1 5 0 1 NA FALSE
y 1 4 0 1 NA FALSE
sp 1 8 NA NA NA NA
values x/y
x NULL [x]
y NULL [y]
sp inflatable_partridge,...,institutional_pronghorn Este juego de capas ya se puede graficar con ggplot2.
# graficar
ggplot() +
geom_stars(data = allspsmg) +
coord_equal() +
scale_fill_scico(palette = "davos", direction = -1, name = "Habitat suitability") +
theme_bw() +
facet_wrap("sp", ncol = 4) +
labs(x = "", y = "") +
theme(legend.position = "bottom") +
labs(title = "")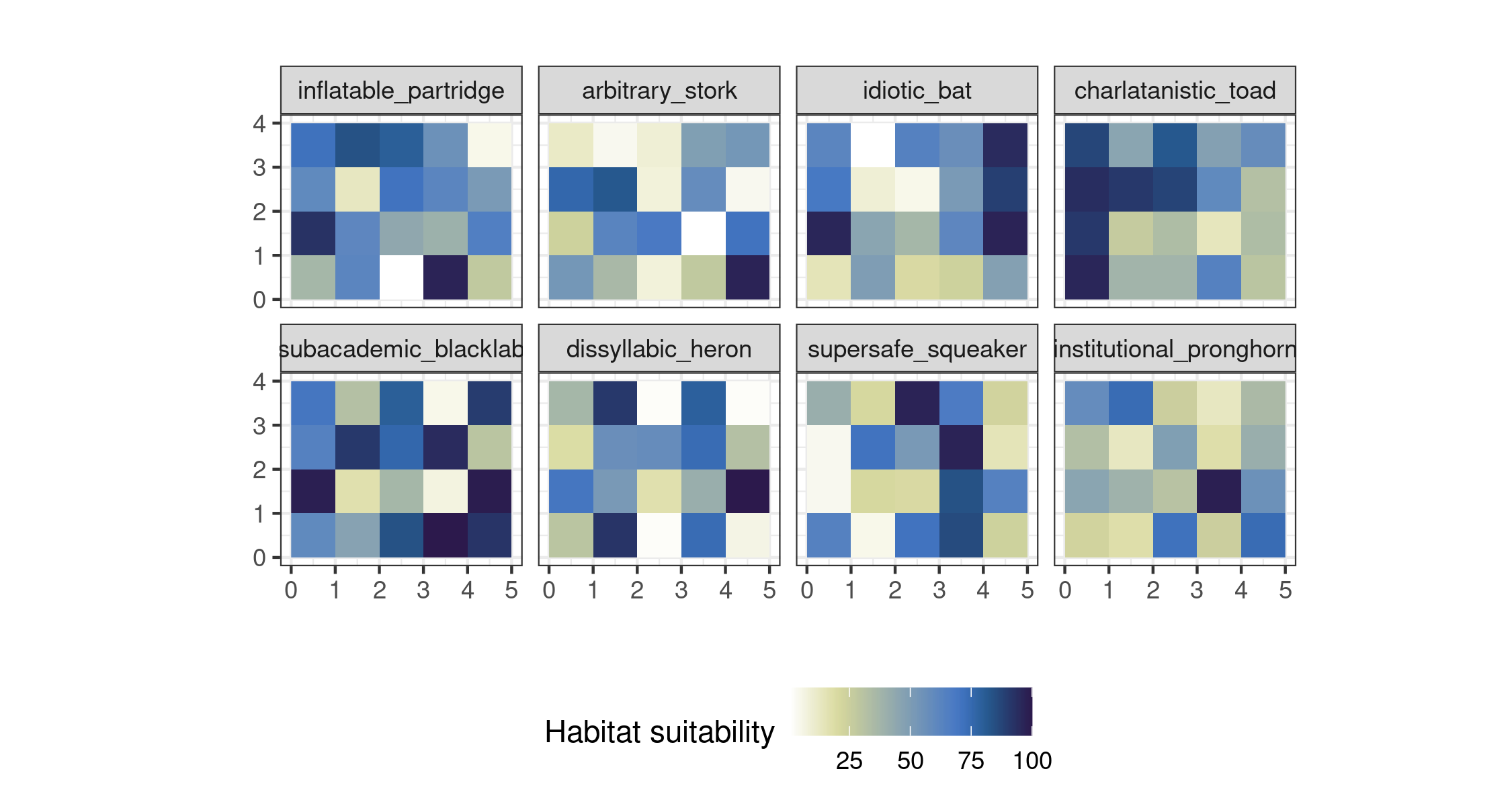
Para re-clasificar todas las capas, podemos usar varios métodos del tidyverse. Aquí vamos a considerar que todos los pixeles con un valor mayor a 70 son presencia, y los demás serían ausencias. Está muy buena la implementación que nos permite usar funciones de dplyr directamente en los atributos del objeto stars.
# pasar a binario
allspRC <-
allspsmg %>%
mutate(presence = case_when(
prob > 70 ~ 1,
TRUE ~ 0
)) %>%
select(presence)Estas reclasificaciones se pueden graficar en un ggplot2 multi-panel.
ggplot() +
geom_stars(data = allspRC) +
coord_equal() +
scico::scale_fill_scico(palette = "davos", direction = -1, name = "Habitat suitability", guide = FALSE) +
theme_bw() +
facet_wrap("sp", ncol = 4) +
labs(x = "", y = "") +
labs(title = "Reclassified presence/absence in grid cells")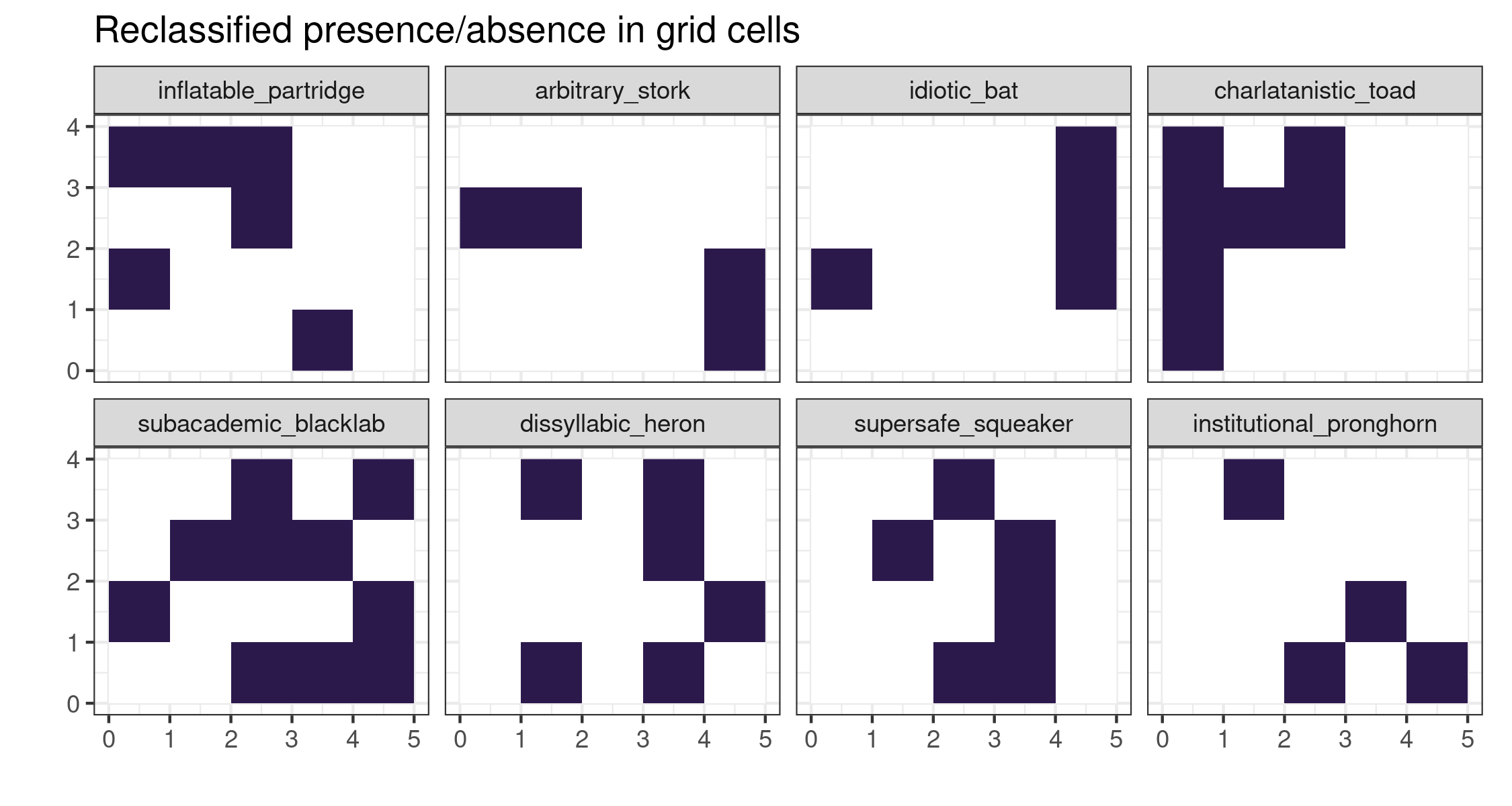
Para sumar las dimensiones, usamos st_apply, que aplica la función sum a las dimensiones con las coordenadas, y así obtenemos el total para cada pixel.
# sumar los valores de celda
cllspRich <-
allspRC %>%
st_apply(c("x", "y"), sum, na.rm = TRUE) %>%
mutate(richness = if_else(sum == 0, NA_real_, sum)) %>%
select(richness)Así queda la capa de riqueza, que ya se puede visualizar. Las sumas tienen sentido, pues los valores en esta capa sí representan la suma de todas las capas de 0/1.
# raster de riqueza
ggplot() +
geom_stars(data = cllspRich) +
coord_equal() +
scale_fill_scico(palette = "davos", direction = -1, name = "Species Richness") +
theme_bw() +
labs(x = "", y = "")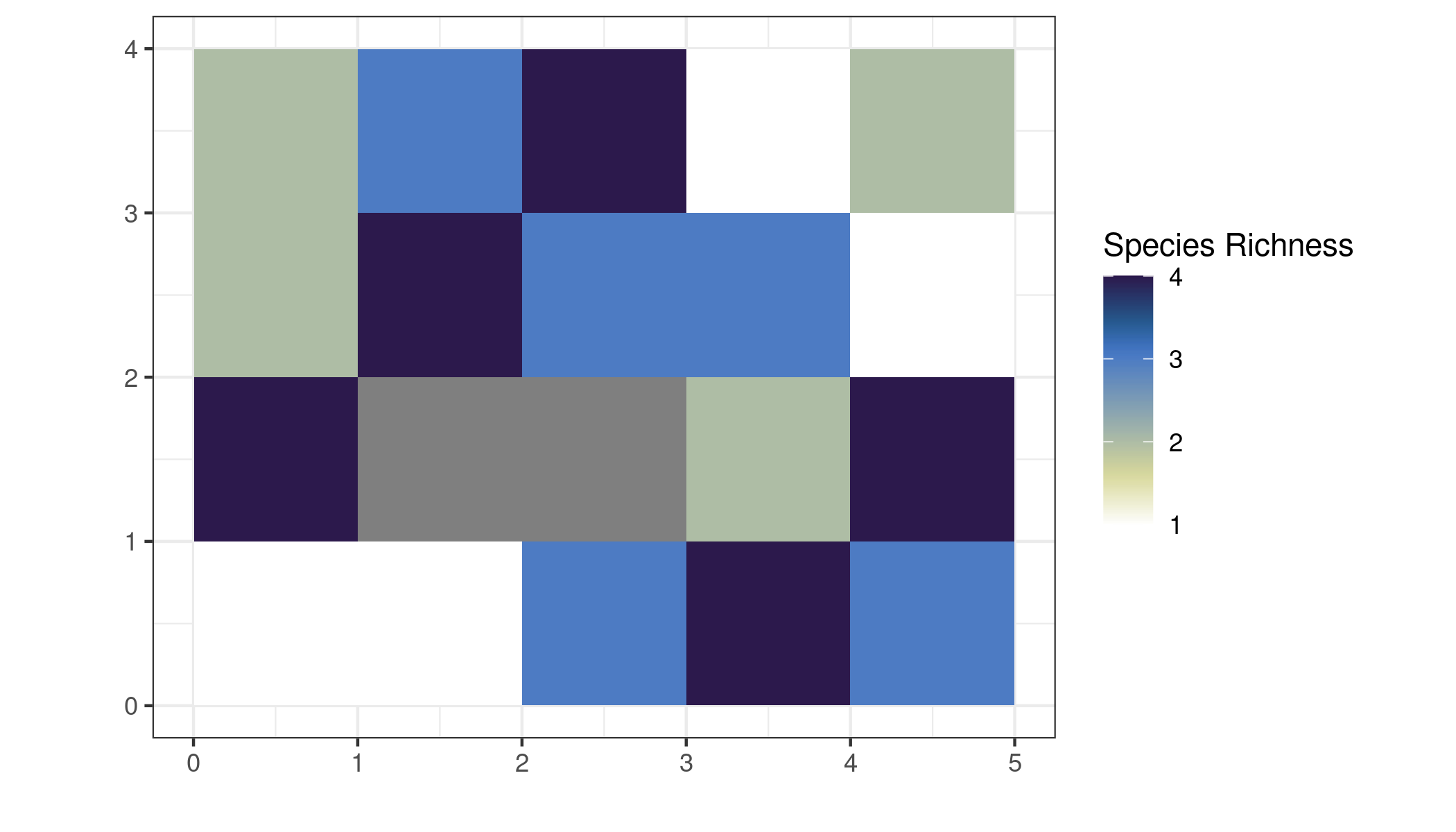
Podemos visualizar estas capas apiladas en 3d, siguiendo esta propuesta de Lauren O’Brien. Pasamos los rasters a vectores (objetos sf), los reestructuramos, y así podemos deformar las geometrías con mutate de dplyr, para darles perspectiva.
# deformando la geometría
allspmgsf <- allspsmg %>%
split() %>%
st_as_sf()
sppolys <- allspmgsf %>% gather("sp", "val", -geometry)
allspRCsf <- allspRC %>%
split() %>%
st_as_sf()
RCsppolys <- allspRCsf %>% gather("sp", "presence", -geometry)
# shear matric
sm <- matrix(c(2, 1.2, 0, 1), 2, 2)
# modifica los polígonos
sppolys_tilt <- sppolys %>% mutate(geometry = geometry * sm)
RCsppolys_tilt <- RCsppolys %>% mutate(geometry = geometry * sm)Podemos acomodar las capas originales y reclasificadas con patchwork
# 3d plot
sppolys_tilt %>%
ggplot() +
geom_sf(aes(fill = val)) +
facet_wrap(~sp, ncol = 1) +
scale_fill_scico(
palette = "davos", direction = -1, name = "Habitat suitability",
guide = guide_colorbar(title.position = "top", label.position = "bottom")
) +
theme_void() +
theme(legend.position = "bottom") +
RCsppolys_tilt %>%
ggplot() +
geom_sf(aes(fill = presence)) +
facet_wrap(~sp, ncol = 1) +
scale_fill_scico(
palette = "davos", direction = -1,
breaks = c(0, 1), labels = c(0, 1),
guide = guide_legend(
title = "presence/absence",
direction = "horizontal",
title.position = "top",
label.position = "bottom",
label.hjust = 0.5,
label.vjust = 1
)
) +
theme_void() +
theme(legend.position = "bottom")
Aplicación con archivos de MaxEnt
Ahora podemos repetir todo el proceso con archivos reales, generados por MaxEnt. Este ejemplo es con modelos de idoneidad ambiental para 96 especies de aves migratorias en Norteamérica. Fueron generados por Diana Stralberg y están archivados en Zenodo.
Primero, descargamos una división política con rnaturalearth.
# división política
divpol <- rnaturalearth::ne_download(scale = "large", type = "countries", returnclass = "sf")
# reproject
divpolLamb <- st_transform(divpol, "+proj=lcc +lat_1=49 +lat_2=77 +lat_0=0 +lon_0=-95 +x_0=0 +y_0=0 +ellps=GRS80 +units=m +no_defs")
# solo Norteamérica
northam <- divpolLamb %>%
filter(stringr::str_detect(SOVEREIGNT, "Canada|United States of America")) %>%
filter(SUBREGION == "Northern America") %>%
select(SOVEREIGNT)Después usé fs y purrr para importar todos los archivos asci desde una carpeta en mi computadora. Para apilarlos, es el mismo procedimiento de arriba.
# modelos, de Stralberg 2012 https://doi.org/10.5281/zenodo.3847272
brbirds <- dir_ls("PATH ON YOUR OWN COMPUTER", regexp = "asc$")
# importar, projectar, y apilar
brlist <- map(brbirds, read_stars)
brlistna <- map(brlist, st_set_crs, "+proj=lcc +lat_1=49 +lat_2=77 +lat_0=0 +lon_0=-95 +x_0=0 +y_0=0 +ellps=GRS80 +units=m +no_defs") # Canada Lambert
breedingbirds <- reduce(brlistna, c) %>%
merge() %>%
st_set_dimensions(names = c("x", "y", "sp"))Delimitamos el área de estudio, y podemos dibujar 8 de estos mapas (elegidos al azar). Aquí sirve slice de dplyr para sacar subconjuntos de un objeto stars.
# definir área de estudio
limsboreal <- st_bbox(st_buffer(st_as_sf(breedingbirds), 20000))
# subconjunto de especies
birds_subset <- breedingbirds %>% slice(sp, sample(1:96, 8))
# mapa
ggplot(northam) +
geom_sf(fill = "#353535", color = "transparent") +
geom_stars(data = birds_subset) +
geom_sf(data = northam, fill = "transparent", size = 0.2, color = "black") +
scale_fill_colormap("Habitat Suitability", na.value = "transparent", colormap = colormaps$portland) +
ggthemes::theme_hc() +
labs(x = "", y = "") +
theme(
panel.background = element_rect(fill = "#577399"),
panel.border = element_rect(colour = "black", fill = "transparent"),
legend.position = "bottom",
panel.grid = element_line(size = 0.08)
) +
coord_sf(
xlim = c(limsboreal["xmin"], limsboreal["xmax"]),
ylim = c(limsboreal["ymin"], limsboreal["ymax"])
) +
facet_wrap(~sp, nrow = 2)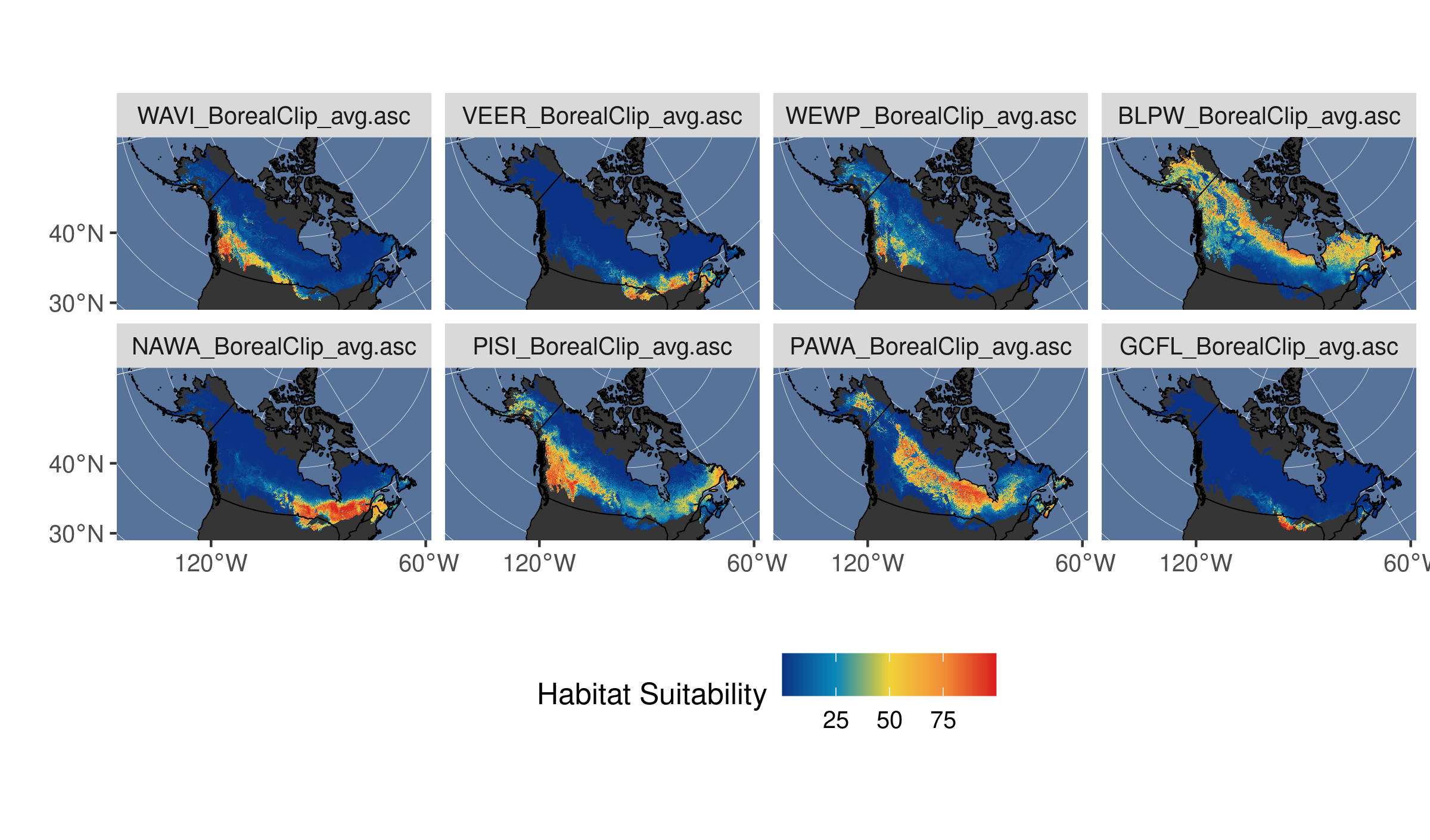
La riqueza de especies se calcula igual que en el ejemplo anterior.
# reclasificar y sumar
breeding_birds_rich <-
breedingbirds %>%
mutate(presence = case_when(
X > 70 ~ 1,
TRUE ~ 0
)) %>%
select(presence) %>%
st_apply(c("x", "y"), sum, na.rm = TRUE) %>%
mutate(richness = if_else(sum == 0, NA_real_, sum)) %>%
select(richness)Ahora ya podemos generar un mapa de riqueza de especies con todas las 96 capas.
# mapa de riqueza de especies
ggplot(northam) +
geom_sf(fill = "#353535", color = "transparent") +
geom_stars(data = breeding_birds_rich) +
geom_sf(data = northam, fill = "transparent", size = 0.2, color = "black") +
scico::scale_fill_scico(
na.value = "transparent", palette = "lajolla", name = "Species richness",
breaks = pretty(1:30),
guide = guide_legend(
title.position = "top",
direction = "horizontal", nrow = 1,
title.theme = element_text(size = 8, face = "bold"),
label.theme = element_text(face = "bold")
)
) +
ggthemes::theme_hc(base_family = "Lato") +
labs(x = "", y = "") +
theme(
panel.background = element_rect(fill = "#577399"),
panel.border = element_rect(colour = "black", fill = "transparent"),
legend.position = c(0.24, 0.1),
legend.background = with_shadow(element_rect(fill = "#EBEBFF"), sigma = 3),
panel.grid = element_line(size = 0.08)
) +
labs(caption = "Passerine bird species richness, n=96. Breeding season") +
coord_sf(
xlim = c(limsboreal["xmin"], limsboreal["xmax"]),
ylim = c(limsboreal["ymin"], limsboreal["ymax"])
)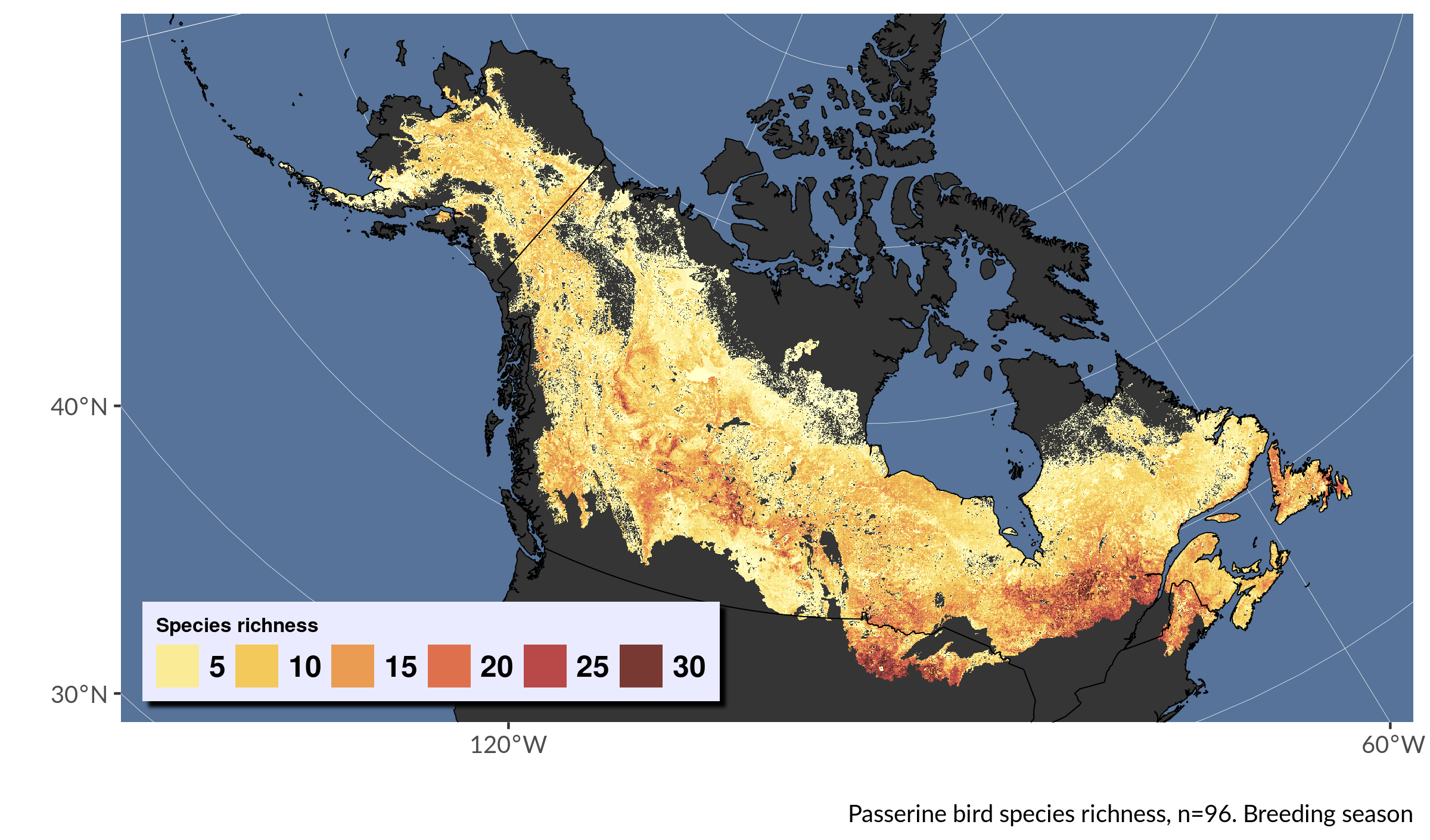
Quedó nítido.
Eso es todo, si tienen preguntas o se traban, no duden en contactarme. Salu-2
