Riqueza de especies usando R y sf
This post is available in English here.
Riqueza de especies y otros procesos espaciales en R
Actualizado en enero de 2020. Había un error que sobreestimaba la riqueza en celdas vacías, y se añade un ejemplo con datos reales.
Para calcular y visualizar de manera resumida la riqueza de especies en una determinada región, hay que calcular el número de registros o de polígonos que resumen la distribución de una especie con respecto a los elementos de otra capa espacial (superpuesta sobre la misma). Esta es una tarea bastante común que se puede hacer con diferentes programas de SIG. He estado trabajando en cuestiones espaciales usando R y el paquete sf, y quería compartir esta forma de cuantificar y graficar mapas de riqueza de especies.
El código en este ejemplo es 100% reproducible, siempre y cuando tengamos instalados los paquetes necesarios. Para este ejemplo escogí trabajar con Costa Rica. Primero vamos a probar este método generando puntos al azar, y lo haremos iterativamente gracias al paquete purrr. Aquí vamos a calcular riqueza de especies usando una cuadrícula y dos diferentes formas de representar la distribución de una especie: con datos puntuales y con polígonos derivados de una envoltura que agrupa a los puntos.
Estos son los pasos principales:
Preparación
- filtrar un subconjunto de datos a partir de un mapamundi para trabajar dentro de un solo país
- generar un número aleatorio de puntos aleatorios dentro del país de estudio para n especies diferentes
- generar envolturas que agrupan a los puntos, y ‘suavizar’ sus esquinas
Análisis espaciales
- generar una cuadrícula que cubra nuestra área de estudio
- hacer una intersección y unión espacial entre las distribuciones de las especies con cada celda de la cuadrícula. ojo con las celdas en las que no hay registros puntuales o sobreposición de polígonos
Mapas
- visualizar la riqueza de especies con colores nítidos, fregones, y adecuados gracias a
ggplot,scicoysfAsí van quedando los mapas con los distintos elementos:
División política:
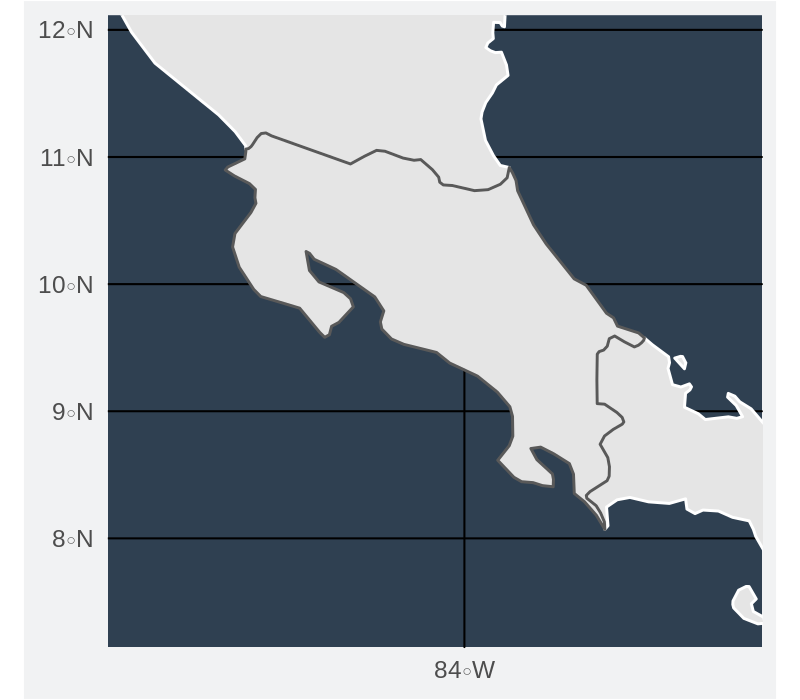
Puntos generados al azar para n especies
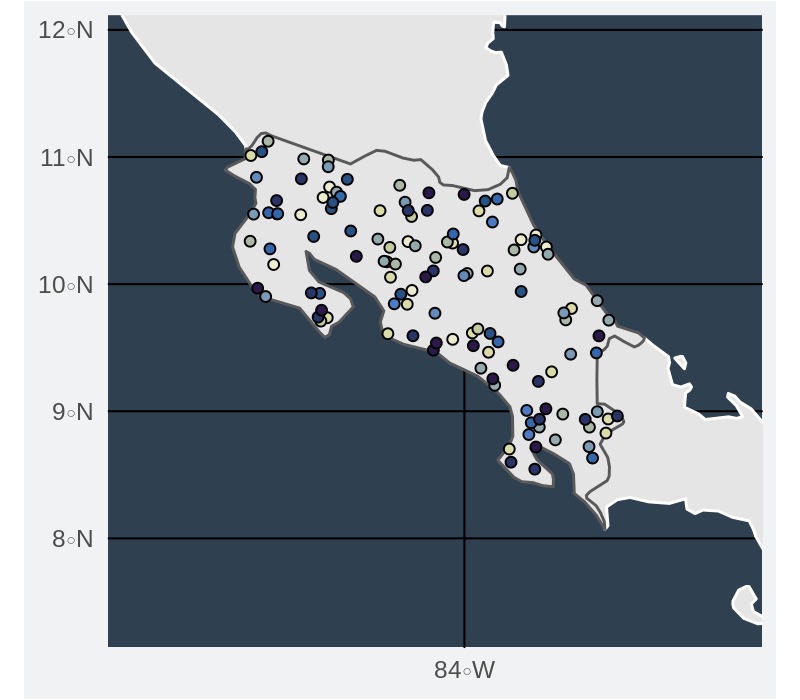
Envolturas
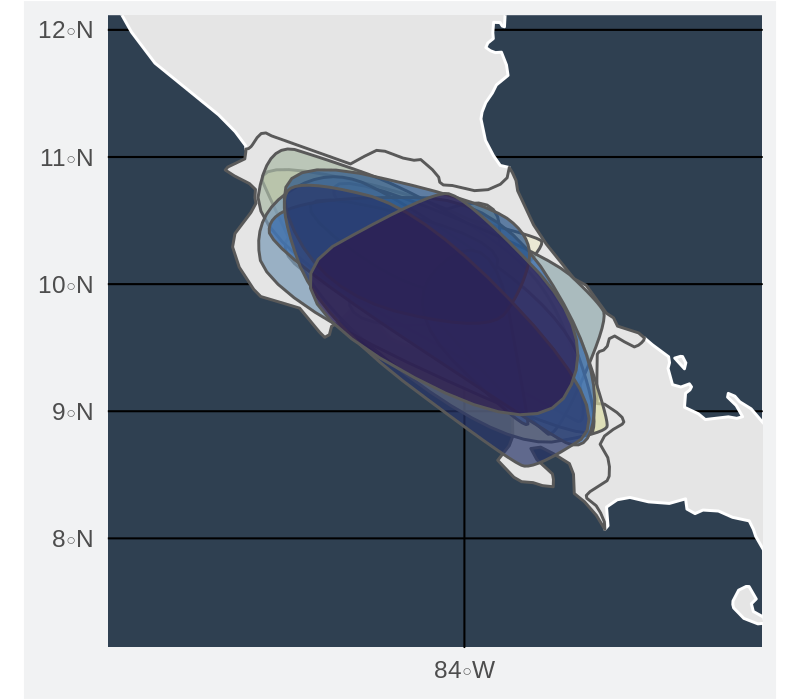
Cuadrícula
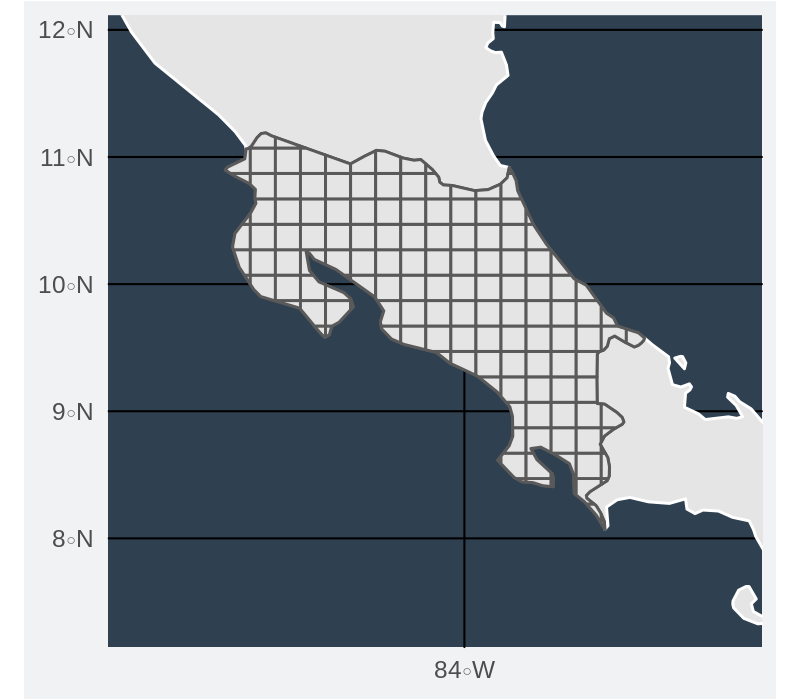
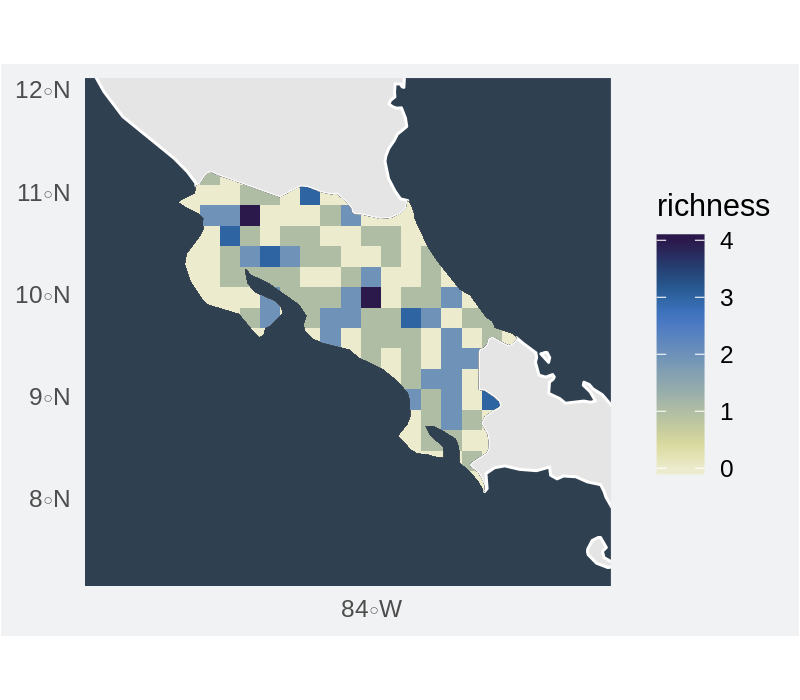
Riqueza de especies calculada a partir de datos puntuales
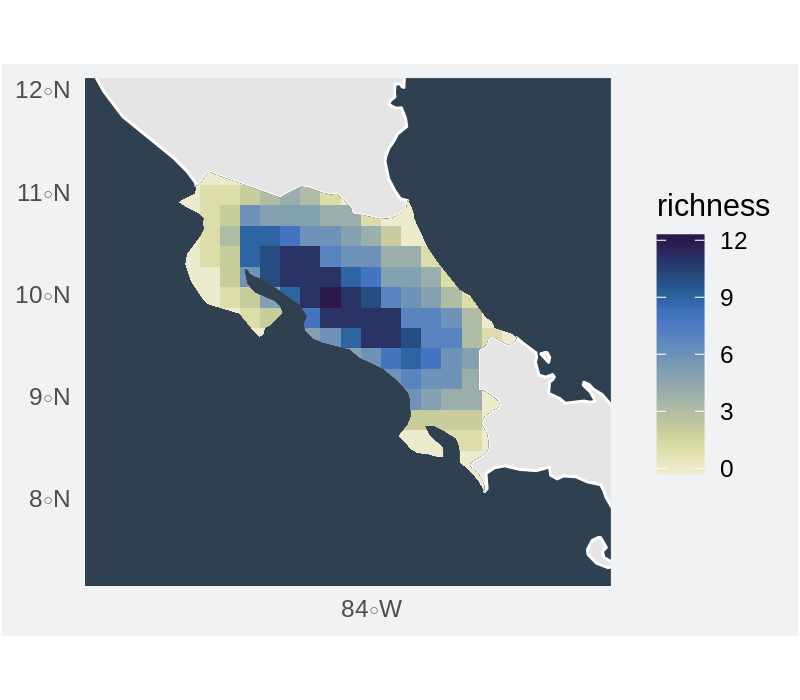
Riqueza de especies calculada a partir de polígonos
Ojo: La función rerun del paquete purrr está muy buena y es un reemplazo útil para no tener que escribir loops.
Al dibujar los mapas, usamos un ‘bounding box’ y la función st_touches para que el mapa quede acotado a nuestra área de estudio y para dibujar los países aledaños, sin necesidad de especificar todo ésto a mano.
Ejemplo con datos reales
Para repetir el ejercicio con datos reales, usamos registros puntuales con coordenadas X y Y que entran en el script como un objeto multipuntos del paquete sf. Los datos son para murciélagos, descargados con el paquete rgbif. A partir de una tabla de tres columnas (especie, longitud, y latitud), agrupamos y resumimos los datos por especie y después generamos un objeto sf con su projección correspondiente (WGS84). El resto de los pasos es igual.
El procedimiento sería el mismo para datos en formato .shp, que se pueden importar fácilmente a sf.
Riqueza de especies de murciélagos en Costa Rica
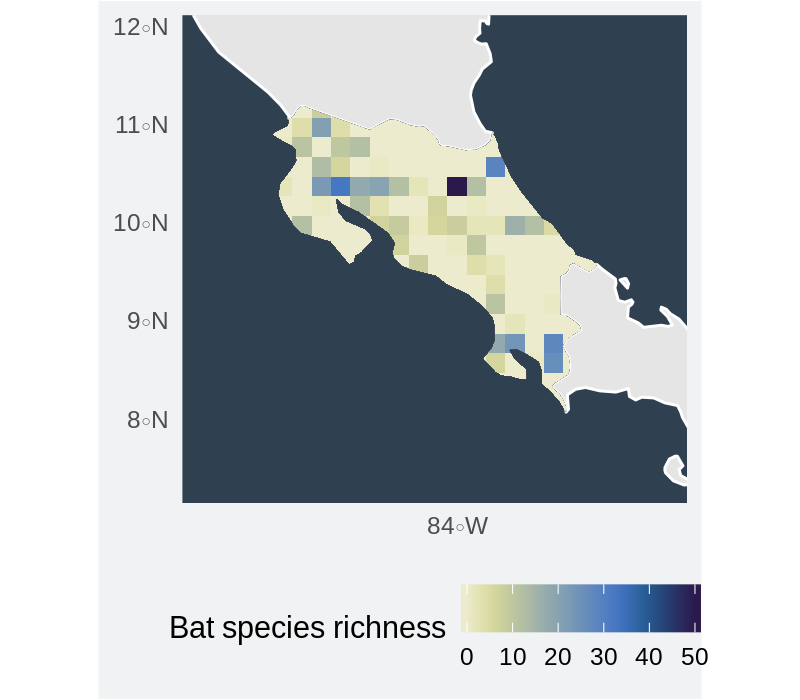
Acepto cualquier duda o sugerencia.
El script:
# cargar paquetes
library(sf)
library(dplyr)
library(ggplot2)
library(scico)
library(rnaturalearth)
library(purrr)
library(smoothr)
library(rgbif)
# mapamundi
worldMap <- ne_countries(scale = "medium", type = "countries", returnclass = "sf")
# subconjunto
CRpoly <- worldMap %>% filter(sovereignt == "Costa Rica")
# puntos al azar, con nombres para los elementos de la lista
sp_occ <- rerun(12, st_sample(CRpoly, sample(3:20, 1)))
names(sp_occ) <- paste0("sp_", letters[1:length(sp_occ)])
# pasar a objeto sf
sflisss <-
purrr::map(sp_occ, st_sf) %>%
map2(., names(.), ~ mutate(.x, id = .y))
sp_occ_sf <- sflisss %>% reduce(rbind)
# convertir a multipunto
sp_occ_sf <- sp_occ_sf %>%
group_by(id) %>%
summarise()
# acotar a nuestra área de estudio
limsCR <- st_buffer(CRpoly, dist = 0.7) %>% st_bbox()
# paises aledaños
adjacentPolys <- st_touches(CRpoly, worldMap)
neighbours <- worldMap %>% slice(pluck(adjacentPolys, 1))
# división política
divpolPlot <-
ggplot() +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly) +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
line = element_blank(),
rect = element_blank()
)
divpolPlot
# puntos
spPointsPlot <-
ggplot() +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly) +
geom_sf(data = sp_occ_sf, aes(fill = id), pch = 21) +
scale_fill_scico_d(palette = "davos", direction = -1, end = 0.9, guide = FALSE) +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
line = element_blank(),
rect = element_blank()
)
spPointsPlot
# envolturas convexas
spEOOs <- st_convex_hull(sp_occ_sf) %>% smooth()
# dibujar envolturas
hullsPlot <-
ggplot() +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly) +
geom_sf(data = spEOOs, aes(fill = id), alpha = 0.7) +
scale_fill_scico_d(palette = "davos", direction = -1, end = 0.9, guide = FALSE) +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
line = element_blank(),
rect = element_blank()
)
hullsPlot
# grid
CRGrid <- CRpoly %>%
st_make_grid(cellsize = 0.2) %>%
st_intersection(CRpoly) %>%
st_cast("MULTIPOLYGON") %>%
st_sf() %>%
mutate(cellid = row_number())
# riqueza en cada celda
richness_grid <- CRGrid %>%
st_join(sp_occ_sf) %>%
mutate(overlap = ifelse(!is.na(id), 1, 0)) %>%
group_by(cellid) %>%
summarize(num_species = sum(overlap))
# riqueza para envoltura
richness_gridEOO <- CRGrid %>%
st_join(spEOOs) %>%
mutate(overlap = ifelse(!is.na(id), 1, 0)) %>%
group_by(cellid) %>%
summarize(num_species = sum(overlap))
# grid en blanco
gridPlot <-
ggplot() +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly) +
geom_sf(data = CRGrid) +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
line = element_blank(),
rect = element_blank()
)
gridPlot
# riqueza
gridRichCR <-
ggplot(richness_grid) +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly, fill = "grey", size = 0.1) +
geom_sf(aes(fill = num_species), color = NA) +
scale_fill_scico(palette = "davos", direction = -1, end = 0.9) +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
line = element_blank(),
rect = element_blank()
) + labs(fill = "richness")
gridRichCR
# riqueza con envolturas
gridRichCR_eoo <-
ggplot(richness_gridEOO) +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly, fill = "grey", size = 0.1) +
geom_sf(aes(fill = num_species), color = NA) +
scale_fill_scico(palette = "davos", direction = -1, end = 0.9) +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
line = element_blank(),
rect = element_blank()
) + labs(fill = "richness")
gridRichCR_eoo
###################################################
# datos de murciélagos
name_suggest(q = "chiroptera")
CRbatsout <- occ_search(
orderKey = 734, country = "CR",
basisOfRecord = "PRESERVED_SPECIMEN", limit = 3000
)$data
# seleccionar especie y coordenadas
CRbatsXY <- CRbatsout %>%
select(species, decimalLongitude, decimalLatitude) %>%
na.omit()
# transformar a objeto sf, especificando las variables con las coordenadas y la projección geográfica
CRbatsXYsf <- st_as_sf(CRbatsXY, coords = c("decimalLongitude", "decimalLatitude"), crs = 4326) %>%
group_by(species) %>%
summarize()
# riqueza
bat_richness_grid <- CRGrid %>%
st_join(CRbatsXYsf) %>%
mutate(overlap = ifelse(!is.na(species), 1, 0)) %>%
group_by(cellid) %>%
summarize(num_species = sum(overlap))
# dibujar
batRichCR <-
ggplot(bat_richness_grid) +
geom_sf(data = neighbours, color = "white") +
geom_sf(data = CRpoly, fill = "grey", size = 0.1) +
geom_sf(aes(fill = num_species), color = NA) +
scale_fill_scico(palette = "davos", direction = -1, end = 0.9, name = "Bat species richness") +
coord_sf(
xlim = c(limsCR["xmin"], limsCR["xmax"]),
ylim = c(limsCR["ymin"], limsCR["ymax"])
) +
scale_x_continuous(breaks = c(-84)) +
theme(
plot.background = element_rect(fill = "#f1f2f3"),
panel.background = element_rect(fill = "#2F4051"),
panel.grid = element_blank(),
legend.position = "bottom",
line = element_blank(),
rect = element_blank()
) + labs(fill = "richness")
batRichCR
