Una versión actualizada (marzo 2020) de esta guía está disponible aquí.
En temas de biodiversidad, es común trabajar con registros puntuales de la distribución de una o más especies, generalmente representadas en coordenadas geográficas xy (longitud/latitud). Para ciertos análisis en macroecología, conservación, y biogeografía, necesitamos datos sobre las condiciones ambientales en cada registro. Estos datos de clima, vegetación, uso de suelo, etc. generalmente existen como capas raster (una representación del área de estudio en formato de matriz dividida en celdas con valores únicos).
Extraer el valor de las celdas en donde ocurren nuestros registros puntuales es fácil usando sistemas de información geográfica (SIG) con interfaces gráficas, que casi siempre tienen algun botón o herramienta dedicada a esta tarea. Para uno de mis proyectos, tuve que extraer los valores de varios raster para los registros puntuales de algunas especies de murciélagos en México. En algún momento decidí que mi regreso a los SIG iba a ser exclusivamente en R, y por eso investigué cómo logar esta extracción de datos sin programas adicionales.
En este caso usé los raster de temperatura y precipitación del proyecto CHELSA Climate. Estos datos se encuentran disponibles desde hace un par de semanas, y parece que tienen mejor exactitud en zonas montañosas que otras opciones existentes como WorldClim. Aquí se pueden descargar los datos de clima en formato geotiff. Son archivos grandes, pero yo no tuve problema para manipularlos en una laptop que ya tiene 5 años.
En este ejemplo, obtuve simultáneamente los valores de dos raster para 150 localidades: la temperatura promedio en enero y la precipitacion promedio en julio. Para reproducirlo, hay que tener los archivos de clima que son Mean january temperature y Mean july precipitation en esta página de descargas, y las coordenadas se pueden leer directamente de este archivo csv.
# cargar paquetes, instalarlos si hace falta usando install.packages()
library(raster)
library(dplyr)
library(sp)
library(rgdal)
# leer los raster de temperatura, en este caso los archivos estan en el directorio de trabajo, que se puede
# averiguar usando getwd()
janTemp <- raster("CHELSA_temp_1_1979-2013.tif") #temperaturas enero
julPrec <- raster("CHELSA_prec_7_1979-2013.tif") # precipitaciones julio
# apilarlos como ingredientes en una hamburguesa
climStack <- stack(janTemp,julPrec)
# leer el archivo con las localidades
localidades <- read.csv("https://raw.githubusercontent.com/luisDVA/codeluis/master/localidades.csv")%>% select(long=2,lat=3)Podemos visualizar las diferentes capas (llamándolas con índices), en este caso usando una extensión geográfica definida con extent y encima de esto se pueden graficar los puntos.
# extension geografica para graficar
# definida por límites máximos y mínimos de long y lat
boxextent <- extent(-115,-90,10,25)
# dibujar el raster y los puntos (en este caso el primer elemento de la pila)
plot(climStack$CHELSA_temp_1_1979.2013,ext=boxextent)
points(localidades) #añade los puntos sobre la gráfica existente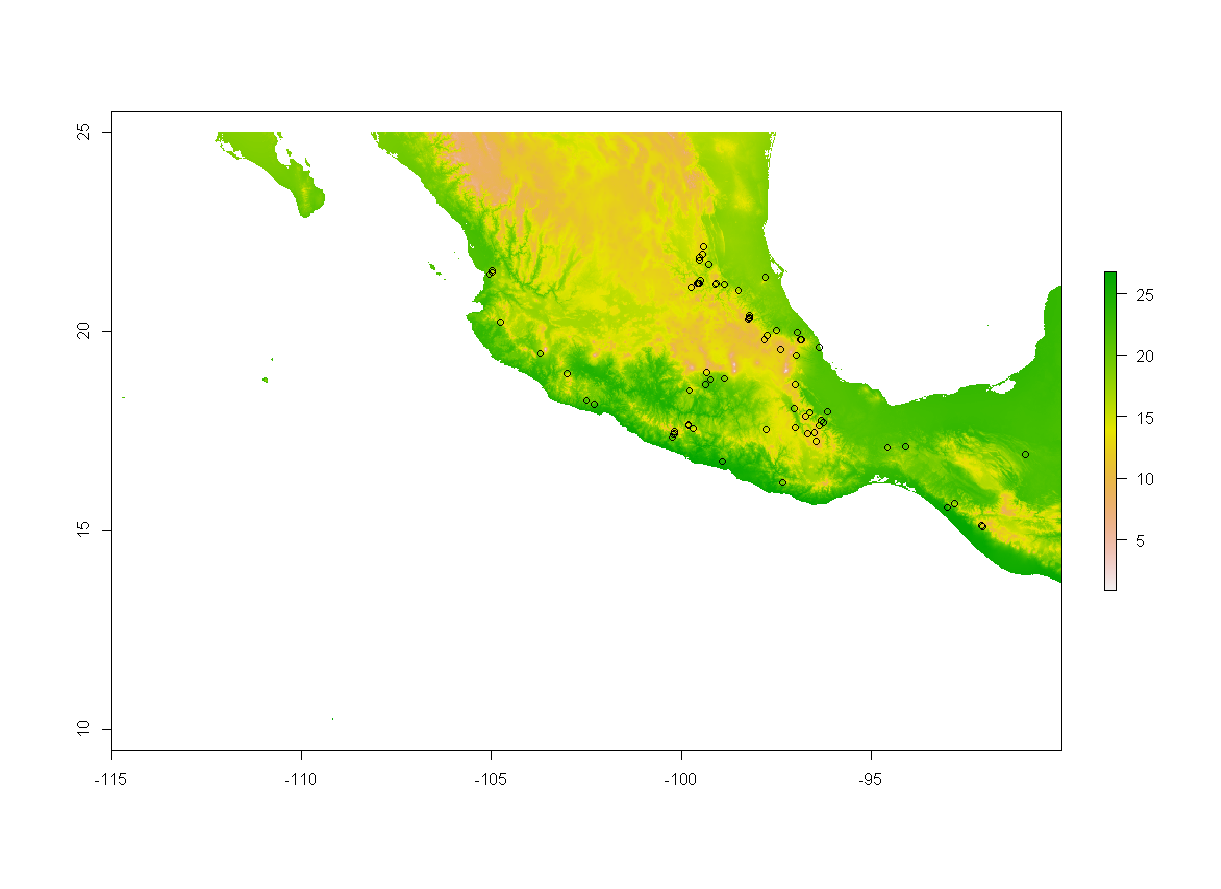
Nota: los conjuntos de capas se pueden recortar con rasterVis::crop por cuestiones de memoria.
La extracción es fácil, y se hace para todos los elementos de la pila al mismo tiempo.
# extrer valores de todas las capas en la pila
extractedClim <- extract(climStack,localidades)
# juntar valores con las coordenadas de las localidades
bioclimat<-cbind(localidades,extractedClim)
# escrbir la tabla en la memoria
write.csv(bioclimat,"biolcimatic.csv")Al final nos queda algo que se ve más o menos así:
| longitude | lat | CHELSA_temp_1_1979-2013 | CHELSA_prec_7_1979-2013 |
|---|---|---|---|
| -96.385 | 19.60055556 | -99.7333 | 17.33361 |
| -96.385 | 19.60055556 | 25.20189667 | 1548.687744 |
| -99.5689 | 21.19917 | 19.83035 | 940.9037 |
| -96.385 | 19.60055556 | 25.20189667 | 1548.687744 |
| -98.7333 | 21.1 | 17.33361 | 545.4098 |
Como tenemos dos variables climáticas, las podemos graficar en 2d como un perfil bioclimático. Este ejemplo es con ggplot pero hay otras opciones para generar gráficas.
library(ggplot2)
ggplot(bioclimat,aes(x=CHELSA_temp_1_1979.2013,y=CHELSA_prec_7_1979.2013))+
geom_point()+geom_density2d()+
xlab("temperatura")+ylab("precipitación")+
theme_minimal()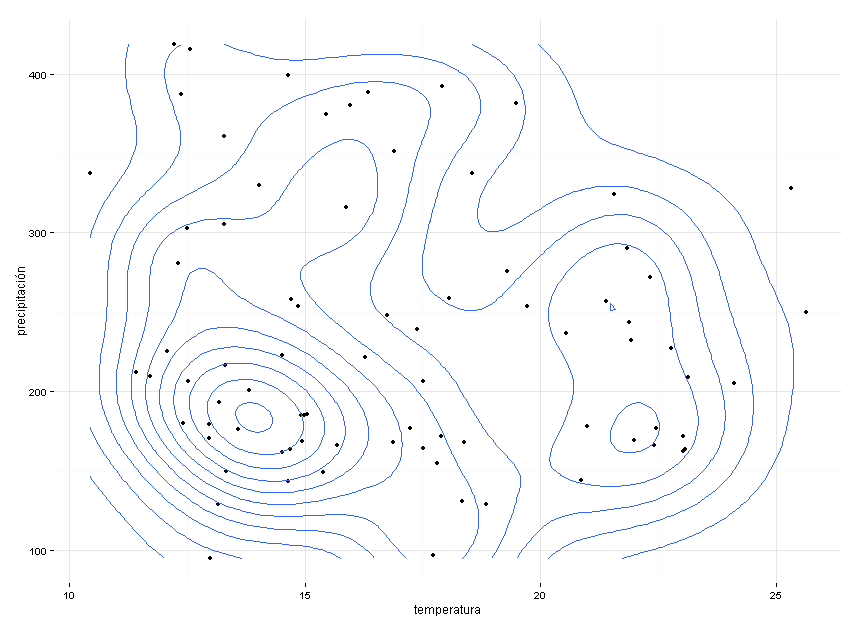
!Listo! Sabiendo extraer datos, los más tardado será la descarga de los raster.
No duden en contactarme si tienen cualquier duda o sugerencia.
