Agrupando puntos en dos dimensiones con ggplot
Actualizado en febrero de 2020 para incluir funciones del paquete
ggforce.
This post is available in English here
Para varios tipos de análisis, es común terminar con gráficas que muestran puntos de dos o más grupos en dos dimensiones. Por ejemplo: análisis de componentes principales, perfiles bioclimáticos, o cualquier combinación de valores en dos ejes. Colaborando en diferentes proyectos, me he encontrado con estas alternativas para agrupar puntos y las quiero compartir en una misma publicación.
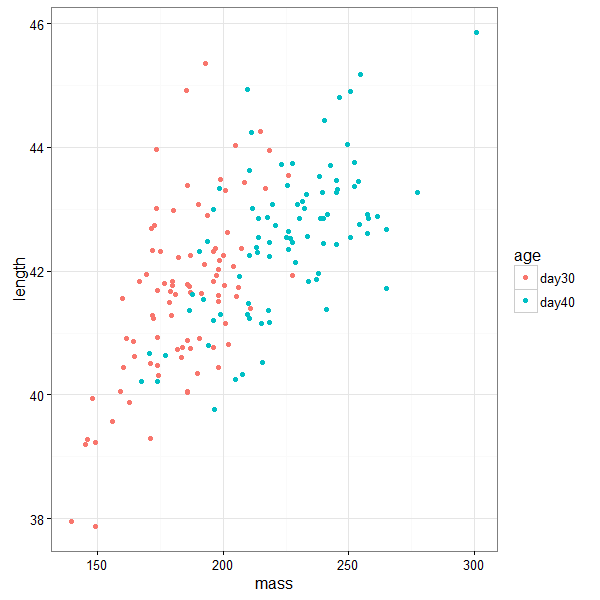
Todos estos métodos son para graficar con ggplot2, y estos ejemplos son con datos reales, obtenidos de la información en línea de este artículo sobre fisiología de codornices. Después de cargar los paquetes requeridos y descargar los datos de Dryad, podemos separar los datos para comparar el peso y el largo de varias aves a los 30 y 40 días de edad.
# cargar paquetes (instalar si es necesario)library(dplyr)
library(ggplot2)
library(ggalt)
library(ggforce)
# descargar datos de Dryad primero, y leer desde nuestro directorio de trabajo
birdData <- read.csv("Morphology data.csv",stringsAsFactors = FALSE)
# separar en dos tablas para 30 y 40 dias de edad
birds40 <- birdData %>% select(mass=Day.40.mass..g.,length=Day.40.head.bill.length..mm.) %>% mutate(age="day40")
birds30 <- birdData %>% select(mass=Day.30.mass..g.,length=Day.30.head.bill.length..mm.) %>% mutate(age="day30")
# juntar en una misma tabla y quitar filas incompletas
birdsAll <- bind_rows(birds30,birds40) %>% na.omit()
# graficar solo los puntos
ggplot(birdsAll,aes(x=mass,y=length,color=age))+geom_point()+theme_bw()
Uno de los métodos más comunes para agrupar puntos es la envoltura convexa (convex hull), que tiene una definición geométrica formal pero que prácticamente es como si rodeáramos al grupo de puntos con una liga elástica. Podemos calcular y dibujar las envolturas para varios grupos usando ggforce.
# graficar con envolturas
ggplot(birdsAll,aes(x=mass,y=length))+
geom_mark_hull(concavity = 5,expand=0,radius=0,aes(fill=age))+
geom_point()+
theme_bw()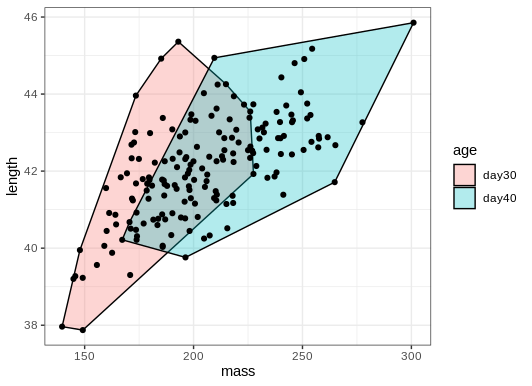
Este tipo de envolturas casi siempre abarcan espacios que no incluyen puntos. Podemos cambiar algunos parámetros de la función para dibujar envolturas más estrechas y con márgenes redondeados.
# envoltura cóncava
ggplot(birdsAll,aes(x=mass,y=length))+
geom_mark_hull(expand=0.01,aes(fill=age))+
geom_point()+
theme_bw()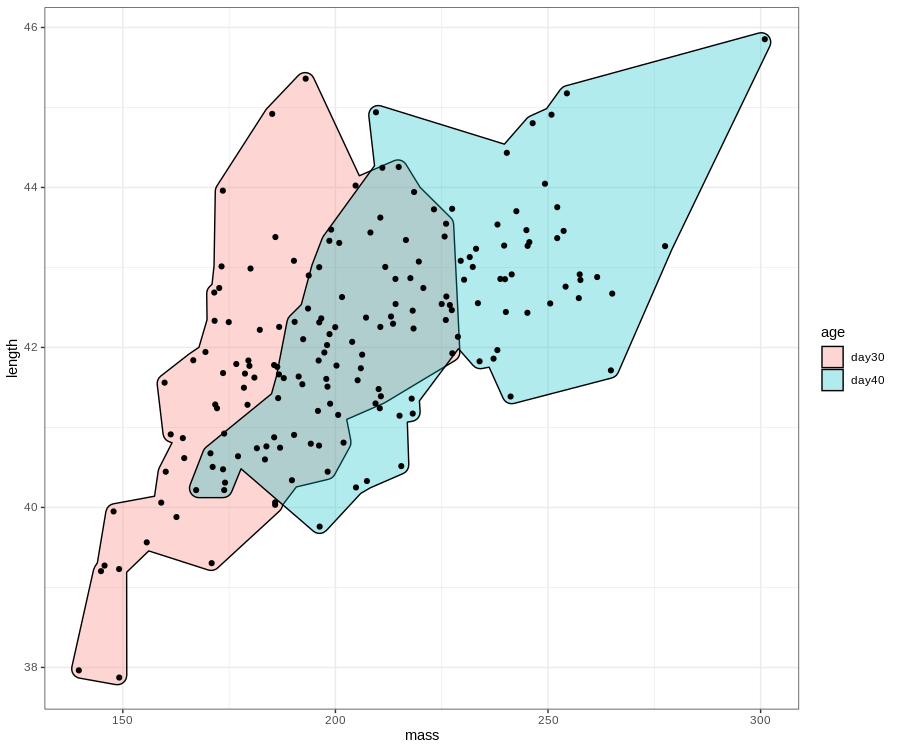
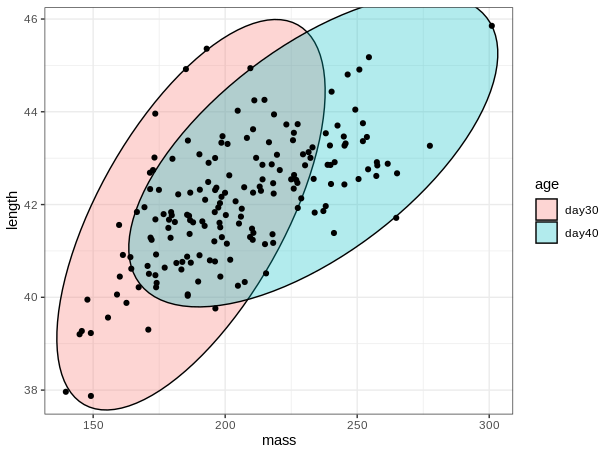
Otra opción muy común es la de agrupar puntos usando elipses. ggforce puede calclular y trazar elipses, que además hereda los parámetros gráficos necesarios para dibujar los colores y la leyenda sin necesidad de especificarlos por separado.
# graficar con elipse
ggplot(birdsAll,aes(x=mass,y=length))+
geom_mark_ellipse(expand = 0,aes(fill=age))+
geom_point()+
theme_bw()
Esta otra opción es la que yo terminé utilizando en mis figuras. Se trata de geom_encircle, una geometría adicional que es parte del paquete ggalt. Este método utiliza curvas diferenciables definidas en porciones mediante polinomios, y termina dibujando polígonos redondeados que se ven bastante bien. Este método es más que nada para agrupar puntos y destacarlos visualmente, y no necesariamente para hacer otros análisis basados en el área del polígono (como ocurre en el caso de las envolturas convexas).
# graficar con poligonos redondeados
ggplot(birdsAll,aes(x=mass,y=length,color=age))+geom_point()+
geom_encircle(expand=0)+ theme_bw()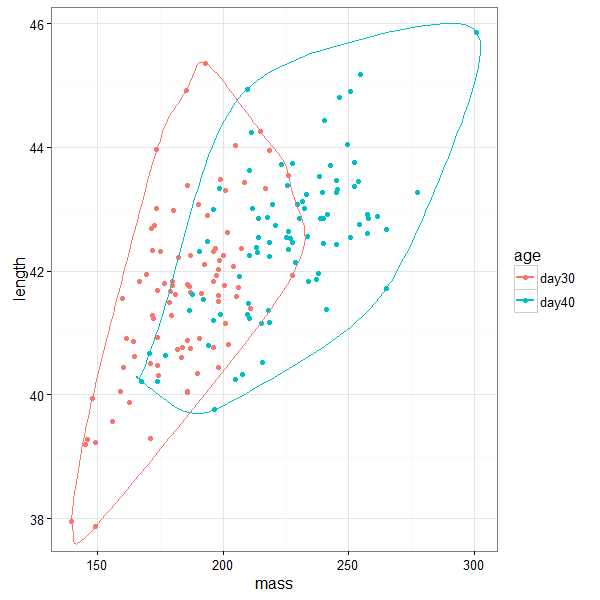
Para estas cuato opciones es posible cambiar los parámetros de relleno y transparencia, estos ajustes sirven para destacar la sobreposición entre grupos.
Espero que les sirva. LD
