Valore máximos y mínimos para datos agrupados
This post is available in English here
Muchas veces tenemos tablas o bases de datos con valores que corresponden a diferentes grupos, sobre todo cuando estamos trabajando con datos de biodiversidad que se encuentran agrupados geográficamente o de acuerdo a alguna taxonomía.
En estos casos es común que necesitemos obtener el valor máximo o mínimo dentro de cada grupo. Por ejemplo:
- las especies de mayor o menor tamaño dentro de cada orden o familia
- los grupos taxonómicos mejor representados en una colección científica, agrupados por estado o municipio
Aquí presento una manera de hacer ésto usando R y la capacidad del paquete dplyr para trabajar con grupos, a través de la función top_n, que combina las funciones filter y min_rank para encontrar los primeros n o los últimos n valores dentro de cada grupo.
Este ejemplo usa datos de la masa cerebral de cientos de mamíferos de este artículo de Gonzalez Voyer et al. En este caso identificamos los valores de masa cerebal máximos y mínimos dentro de cada grupo taxonómico (órdenes en este ejemplo).
Primero podemos descargar los datos directamente desde Dryad y revisar su estructura. Como queremos valores máximos y mínimos, podemos quitar aquellos órdenes que son monotípicos o que sólo tienen datos para una sola especie en esta base de datos.
# cargar los paquetes necesarios (hay que instalarlos primero)
library(dplyr)
library(ggplot2)
# descargar los datos
brains <- read.csv("http://datadryad.org/bitstream/handle/10255/dryad.114692/Gonzalez-Voyer_et_al_Evolution_Brain_Data.csv?sequence=1")
# revisarlos
glimpse(brains)
# contar cuántas especies hay en cada orden
brains %>% count(Taxonomic_order)
# tabla con ordenes que tienen 2 ó más especies
brains2 <- brains %>% group_by(Taxonomic_order) %>% filter(n()>1)Ahora podemos usar la función de agrupamiento group_by y luego top_n pare obtener los valores máximos y mínimos dentro de cada grupo. En este caso obtuve estos valores por separado y luego los junté en la misma tabla usando bind_rows.
# tabla con los valores máximos
brtop <- brains2 %>% group_by(Taxonomic_order) %>% top_n(1,Brain_mass_g)
# tabla de valores mínimos
## se utiliza la misma función pero con un índice negativo
brbottom <- brains2 %>% group_by(Taxonomic_order) %>% top_n(-1,Brain_mass_g)
# juntar y acomodar
minmaxBr <- bind_rows(brtop,brbottom)
minmaxBr <- arrange(minmaxBr,Taxonomic_Order)
# revisar los resultados para murciélagos
minmaxBr %>% filter(Taxonomic_order=="Chiroptera")| Taxonomic_order | Species_name | Brain_mass_g |
|---|---|---|
| Chiroptera | Pteropus_giganteus | 7.605096 |
| Chiroptera | Pipistrellus_subflavus | 0.125000 |
Podemos ver que el murciélago (Orden Chiroptera) con la mayor masa cerebral es Pteropus giganteus y el de menor masa cerebral es Pipistrellus subflavus. Podemos graficar estos pares de valores para algunos órdenes para ver la variación.
# graficar
minmaxBr %>% filter(Taxonomic_order %in% sample(levels(minmaxBr$Taxonomic_order),5)) %>%
ggplot(aes(x=Taxonomic_order,y=log(Brain_mass_g)))+geom_path()+theme_minimal()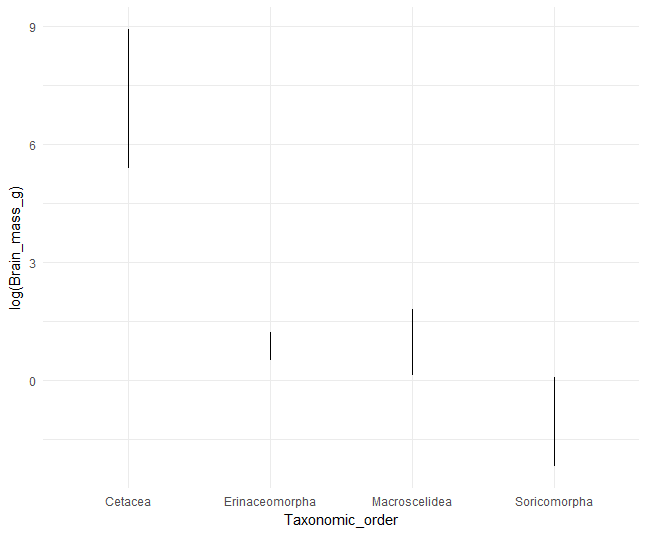
Espero que esta breve guía les sirva. No duden en comunicarse con cualquier duda. Feliz año 2017.

