[1] "((A,B),X);"Filogenias en R
Filogenias en R
Primero, ¿Qué es un árbol Filogenético?
Un árbol filogenético es un diagrama que muestra la relación de parentezco entre especies (o taxones)
Uno de los formatos más utilizados para representar filogenias son el Newick(.tree)
El formato Newick es una notación para representar parentescos utilizando parentesis y comas.
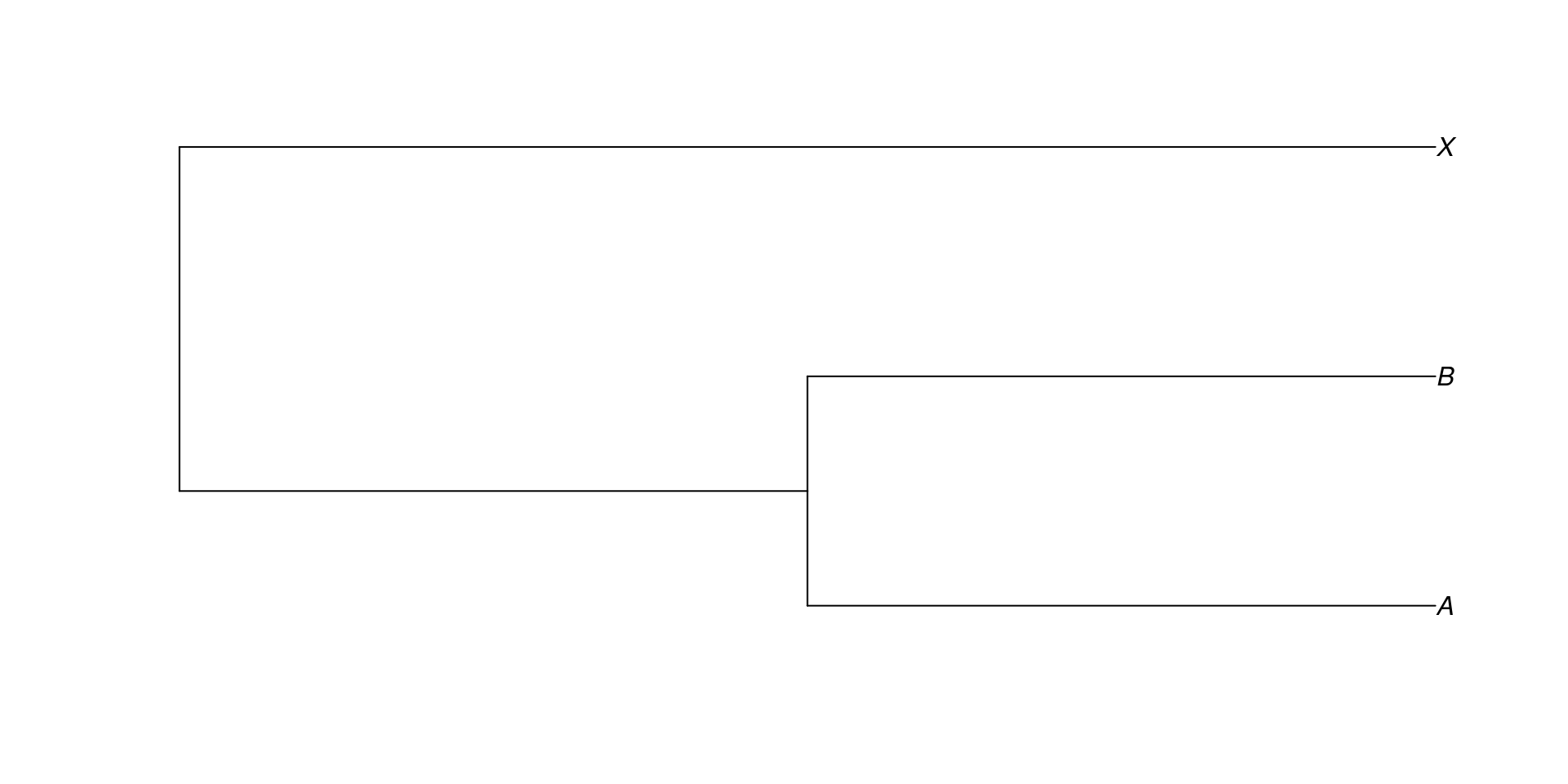
Si queremos decir que A y B se parecen más entre ellos que con X, lo anotaríamos así:
Para que R pueda identificar está notación, se puede usar ape:
El formato Newick es anidado, por lo que podemos explorar la relación entre diferentes grupos. Por ejemplo:
Sabemos que los lobos, los coyotes y los zorros pertenecen a Canidae, pero que los lobos y los coyotes son del mismo género

Y si agregamos murciélagos, todos ellos pertenecen a los mamíferos:

Si quisieramos agregar cuervos y zanates. ¿Estos dónde irían?

Y si agregamos una planta, los mamíferos y las aves se anidarían.

- En este ejercicio haremos uso del paquete ggtree para graficar, personalizar, anotar y en general mejorar árboles filogenéticos
Paquetes necesarios:
En este caso usaremos un árbol filogenético en particular (la filogenia de Icteridae). Sin embargo, estos ejemplos se pueden hacer en su mayoría con cualquier otra filogenía que tengan disponible.
Phylogenetic tree with 100 tips and 99 internal nodes.
Tip labels:
Icterus_melanopsis, Icterus_northropi, Icterus_laudabilis, Icterus_dominicensis, Icterus_cayanensis, Icterus_pyrrhopterus, ...
Node labels:
761, 762, 763, 764, 765, 766, ...
Rooted; includes branch lengths.Veamos esta filogenia graficandola usando ape, la manera base para R.

Ahora graficaremos el mismo árbol utilizando ggtree, el cual sigue una formula idéntica a la de ggplot. Nuestro árbol base se llamará p1:

Con p1, al igual que con cualquier otro gráfico de ggplot, podemos agregar parametros de graficación para personalizar o agregar anotaciones a nuestra filogenia. Primero empezaremos con los parametros de personalización. Por ejemplo, podemos cambiar la disposición de la filogenia utilizando el parametro layout_:

También podemos cambiar el color del fondo del gráfico

Agregar las etiquetas para las puntas de una manera personalizable:

formas a las puntas:

Etiquetas a los nodos:

o formas a los nodos:

e incluso agregar una escala temporal a la filogenia, con la ayuda del paquete deeptime

Cómo en ggplot, todos estos parámetros son aditivos, por lo cual puedes construir la filogenía de manera gradual

Ahora para la anotación de los árboles, se pueden hacer varias cosas, a mi, por ejemplo, me gusta mucho el género Quiscalus, y quisiera saber donde se encuentra en la filogenia.
Para esto primero debemos encontrar el nodo del ancestro en común más reciente para este grupo. Usando un tibble y la estructura de dplyr es muy facil, primero transformamos nuestro árbol en un tibble y despues utilizando un filtro buscamos las especies de Quiscalus:
# A tibble: 6 × 4
parent node branch.length label
<int> <int> <dbl> <chr>
1 146 41 1.82 Quiscalus_quiscula
2 148 42 0.851 Quiscalus_niger
3 149 43 0.335 Quiscalus_major
4 149 44 0.335 Quiscalus_mexicanus
5 150 45 1.02 Quiscalus_nicaraguensis
6 150 46 1.02 Quiscalus_lugubris Después usando la función MRCA podemos encontrar el ancestro en común de estas especies, en las cuales podemos usar los nombres de las especie con la longitud de rama más alargada y con la longitud de rama más corta, o sus nodos:
# A tibble: 1 × 4
parent node branch.length label
<int> <int> <dbl> <chr>
1 144 146 1.37 806 Ahora sabiendo que el ancestro en común de Quiscalus se encuentra en el nodo 146, puedo utilizar esta información para anotar la filogenia utilizando el parametro geom_cladelab:

y que tal el genero que tiene el nombre de la familia: Icterus:
# A tibble: 31 × 4
parent node branch.length label
<int> <int> <dbl> <chr>
1 111 1 0.193 Icterus_melanopsis
2 111 2 0.193 Icterus_northropi
3 114 3 1.47 Icterus_laudabilis
4 114 4 1.47 Icterus_dominicensis
5 116 5 0.197 Icterus_cayanensis
6 116 6 0.197 Icterus_pyrrhopterus
7 115 7 0.943 Icterus_auricapillus
8 117 8 1.49 Icterus_bonana
9 117 9 1.49 Icterus_portoricensis
10 118 10 2.24 Icterus_prosthemelas
# ℹ 21 more rows
También podemos dibujar una linea entre dos taxa, que pudieran o no estár relacionados, utilizando el parametro geom_strip:

Un parametro de anotación muy bueno, también es el geom_highlight, el cual nos permite destacar clados en particular, utilizando los nodos de ancestro en común:

¿Qué clado es este?

Una función bastante interesante de ggtree es que se pueden personalizar las filogenías utilizando recursos en línea como phylopic o enriquecerlas con imagenes propias. Para poder hacer uso de esta función, primero debemos cargar un paquete extra:
Hacer uso de phylopic para personalizar las anotaciones de las filogenias requiere que primero hagamos una tabla con los nodos, el nombre de la especie o clado a los cuales vamos a anotar y el phylopic_id.
En este ejemplo utilizaré los clados Quiscalus y Agelaius, que sé que tienen imagenes indexadas en phylopic. Encontrar los phylopic_id es fácil usando la función phylopic_uid:
name uid
Quiscalus Quiscalus 2ada4e2f-35ab-48d2-b32e-3bad57033dd4
Agelaius Agelaius 9a3f70a3-2ea9-45aa-8a1a-7c599952fd6cCon estos ids, ya podemos crear nuestra tabla con los datos necesarios y después gráficar nuestra filogenia:

Además, usando la argumentación como en ggplot, podemos personalizar los colores de nuestros phylopics:

Utilizando estas parametrizaciones, podemos crear una filogenia bastante atractiva:
pr<-p1+ geom_cladelab(data = dt,
mapping = aes(node = node, label = genus,
image = image, color = genus),
geom = "phylopic", offset = 0, offset.text=0.5)+
scale_color_manual(values=c("#f75419","purple"))+
geom_highlight(node=146,alpha=0.5,fill="purple",type = "rect")+
geom_highlight(node=136,alpha=0.5,fill="#f75419",type = "rect")
pr
y este es un ejemplo de el tipo de filogenias que pueden crearse haciendo uso de todas estas parametrizaciones:
tree<-read.tree(here("data","nodedtree.txt"))
nodes<-c(825,725,921,1042,1080,1375,707)
labels<-c("Parulidae","Icteridae","Passerellidae","Cardinalidae","Thraupidae","Emberizidae","Calcariidae")
#
iu2<-phylopic_uid(c("Setophaga","Quiscalus","Passerellidae","Cardinalis","Thraupidae","Emberizidae","Emberiza"),seed=1)
dt<-data.frame(node=nodes,name=labels,image=iu2$uid)
p2<-ggtree(tree,layout="circular",color="white")+theme_tree("black")
p3<-p2+
geom_highlight(node=825,fill="yellow")+
geom_cladelab(node=825,label="Parulidae",barcolor="yellow",textcolor="white",offset.text=4, fontsize=4)+
geom_highlight(node=725,fill="orange")+
geom_cladelab(node=725,label="Icteridae",barcolor="orange",textcolor="white",offset.text=5, fontsize=4,angle=45)+
geom_highlight(node=921,fill="brown")+
geom_cladelab(node=921,label="Passerellidae",barcolor="brown",textcolor="white",offset.text=1, fontsize=4)+
geom_highlight(node=1042,fill="red")+
geom_cladelab(node=1042,label="Cardinalidae",barcolor="red",textcolor="white",offset.text=7, fontsize=4,angle=-45,align=T)+
geom_highlight(node=1079,fill="lightgreen")+
geom_cladelab(node=1079,label="Thraupidae",barcolor="lightgreen",textcolor="white",offset.text=2, fontsize=4)+
geom_highlight(node=1375,fill="magenta")+
geom_cladelab(node=1375,label="Emberizidae",barcolor="magenta",textcolor="white",offset.text=1, fontsize=4,align=T)+
geom_highlight(node=707,fill="blue")+
geom_cladelab(node=707,label="Calcariidae",barcolor="blue",textcolor="white",offset.text=1, fontsize=4,align=T)
Para poder anotar las filogenias con imagenes, es recomendable usar árboles filogéticos pequeños, en los cuales quizá con un grupo de representantes bastaría. En este ejemplo usaremos una filogenia parafiletica con los generos Quiscalus (Los zanates), Icterus (Las calandrias), Molothrus (Los tordos), Agelaius (Los sargentos) y Psarocolius (Las oropendolas):
Entonces, primero recuperamos un representante de cada grupo, ape es muy bueno para esto usando la función keep.tip:

Una vez teniendo este árbol parafilético, podemos colocar las imagenes gusto en sus grupos correspondientes, es importante considerar, que las imagenes deben tener el nombre exacto del grupo o especie y el mismo formato:

También es posible agregar especies a la filogenia dentro de sus respectivos generos, pero hay que tener cuidado con esto, pues posiblemente nos genere politomías o colocarlas de manera discordante, y esto puede ser problématico para estudios de diversificación o evolución de atributos.
Volveremos a usar nuestra filogenia de grupos y le agregaremos dos especies de Icterus y una especie de Quiscalus

Para agregar las especies, utilizamos la función add.species.to.genus de phytools:

Finalmente, se puede usar ggtree, para graficar atributos de las especies en la filogenia.
Para hacer esto, primero debemos cargar los atributos de los Icteridos, en este caso usaremos el Hand Wing Index (HWI) y el hábito migratorio:
Una vez cargados los datos, la manera más fácil de utilizarlos es uniendolos a la filogenia usando la funcion full_join, es importante que las especies estén etiquetadas como label, para que la función las reconozca:
names(hwi)<-c("label","hwi")
names(migrants)<-c("label","migratory")
hwimigrants<-hwi%>%
left_join(migrants,by="label")
datatree<-full_join(ictree,hwimigrants,by="label")
datatree'treedata' S4 object'.
...@ phylo:
Phylogenetic tree with 100 tips and 99 internal nodes.
Tip labels:
Icterus_melanopsis, Icterus_northropi, Icterus_laudabilis,
Icterus_dominicensis, Icterus_cayanensis, Icterus_pyrrhopterus, ...
Node labels:
761, 762, 763, 764, 765, 766, ...
Rooted; includes branch lengths.
with the following features available:
'', 'hwi', 'migratory'.
# The associated data tibble abstraction: 199 × 5
# The 'node', 'label' and 'isTip' are from the phylo tree.
node label isTip hwi migratory
<int> <chr> <lgl> <dbl> <chr>
1 1 Icterus_melanopsis TRUE 26.5 No-migrant
2 2 Icterus_northropi TRUE 21.0 No-migrant
3 3 Icterus_laudabilis TRUE 22.2 No-migrant
4 4 Icterus_dominicensis TRUE 26.5 No-migrant
5 5 Icterus_cayanensis TRUE 26.3 No-migrant
6 6 Icterus_pyrrhopterus TRUE 17.8 No-migrant
7 7 Icterus_auricapillus TRUE 17.3 No-migrant
8 8 Icterus_bonana TRUE 16.9 No-migrant
9 9 Icterus_portoricensis TRUE 22.3 No-migrant
10 10 Icterus_prosthemelas TRUE 24.1 No-migrant
# ℹ 189 more rows¡Listo! Ahora tenemos una filogenia con atributos y podemos gráficarlos juntos
Primero gráficaremos los valores continuos del HWI sobre las puntas del árbol en una escala de colores:

podemos también personalizar esta escala:

¿Y cómo se verían los datos binarios?

y ¿Pueden combinarse?
Esto puede hacerse con un lenguaje de dplyr
px<- py%<+% migrants + geom_tippoint(aes(color=migratory),shape=15)+
scale_color_manual(values = c("#961d29","#1420a3"))+
scale_fill_manual(values = c("#961d29","#1420a3"))
px+ geom_facet(panel="HWI",data = hwi,geom=geom_col,mapping=aes(x=hwi,color=migratory,fill=migratory),orientation='y')+
theme_tree2()
Pero, ¿Qué pasa si no me sé la taxonomía o no tengo la filogenía del grupo que estoy estudiando?
¿Se acuerdan de esta filogenia?

Haciendo uso de recursos en línea como Open tree of Life(OTL), podemoos obtener una filogenia utilizando las especies que necesitemos.
OTL tiene un útil paquete en R (rotl), que usaremos para obtener la filogenia con la planta, aves y mamiferos.
library("rotl")
otl_tree<-rotl::tnrs_match_names(c("Quiscalus mexicanus","Canis lupus","Corvus corax","Canis latrans","Vulpes vulpes","Desmodus rotundus","Sideroxylon celastrinum")) %>%
pull(ott_id) %>%
tol_induced_subtree(label_format="name")
Progress [---------------------------------] 0/211 ( 0%) ?s
Progress [==============================] 211/211 (100%) 0s

Para la función de rtol, no es necesario introducir las especies en orden, ya que ésta recuperará la taxonomia válida y nos arrojará una filogenia podada.
En este caso, OTL nos regresó exactamente la misma filogenia que creamos al principio:
