Patrones geográficos de riqueza de especies
Áreas de distribución y patrones geográficos de riqueza
“To do science is to search for repeated patterns, not simply to accumulate facts” R. MacArthur
- A partir de las distribuciones geográficas de las especies es posible describir y evaluar diferentes patrones de biodiversidad que emergen cuando estudiamos conjuntos de especies (y no a nivel indivual)
Áreas de distribución y patrones geográficos de riqueza
En este ejercicio veremos cómo usar las distribuciones de las especies (polígonos, como los que generamos en el ejercicio anterior) para generar el patrón geográfico de riqueza de especies
Usaremos dos paquetes (
letsRyepm) para mostrar diferentes maneras de llegar a lo mismo, considerando paquetes obsoletos (y que pronto se retirarán) y nuevos (que suplen los anteriores)
Los datos (polígonos) son de una familia de aves (Passeriformes:Emberizoidae:Icteridae) obtenidos de BirdLife International
Paquetes necesarios:
letsR (Vilela & Villalobos 2015 MEE)
Cargar los polígonos de las especies de interés (shapefiles en su carpeta de trabajo)
OGR data source with driver: ESRI Shapefile
Source: "/home/luisd/Dropbox/darcyDB/PDRF/curso-R-biodiversidad/data/icteridae_maps/icteridae_maps.shp", layer: "icteridae_maps"
with 190 features
It has 26 fieldsCrear una matriz de presencia-ausencia a partir de los polígonos
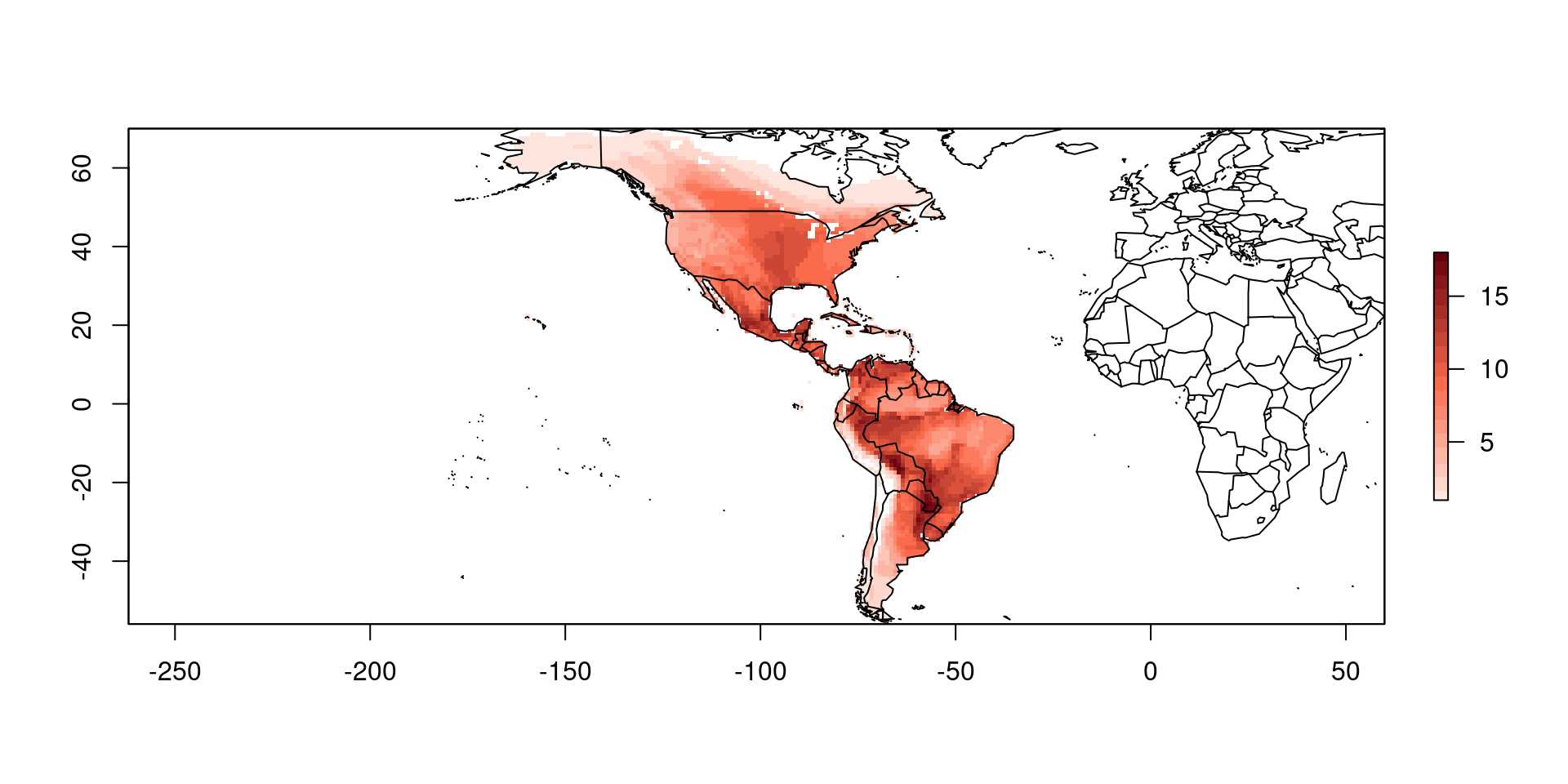
¿Cómo se ve el patrón geográfico?
¿Qué tiene el objeto que se graficó?
Vamos a verlo en ggplot
Primero, convertimos el raster en un data.frame
rdf <- as.data.frame(icteri.pam$Richness_Raster, xy=TRUE) #Convert raster to data.frame
names(rdf)[3] <- 'richness' #Name value column
head(rdf) x y richness
1 -166.5 69.5 0
2 -165.5 69.5 0
3 -164.5 69.5 0
4 -163.5 69.5 0
5 -162.5 69.5 0
6 -161.5 69.5 0 x y richness
1 -166.5 69.5 NA
2 -165.5 69.5 NA
3 -164.5 69.5 NA
4 -163.5 69.5 NA
5 -162.5 69.5 NA
6 -161.5 69.5 NAAhora sí, lo graficamos
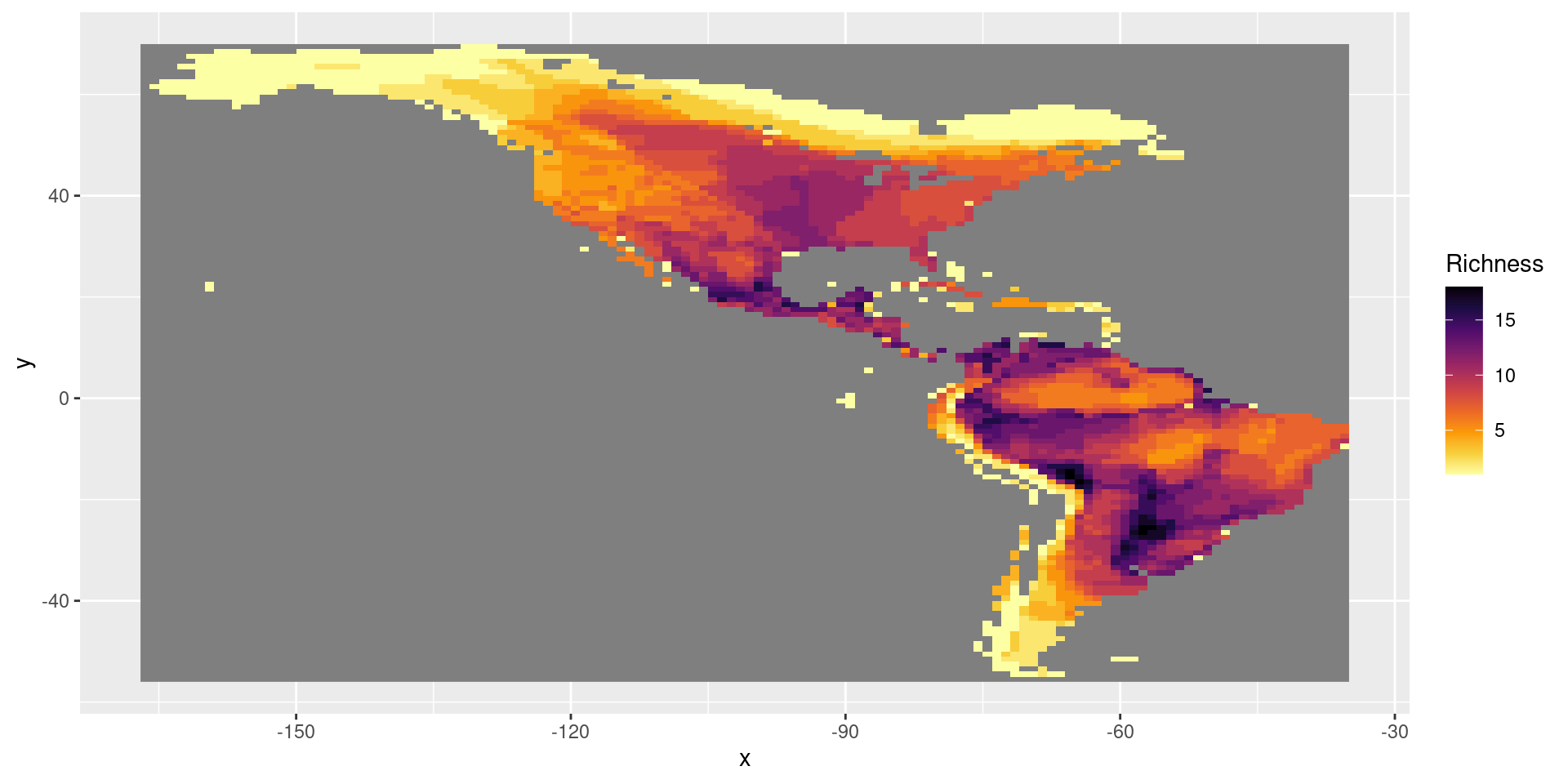
EcoPhyloMapper - epm (Title et al. 2022 MEE)
Cargar nuevamente los polígonos, pero ahora con el paquete ‘nuevo’ y recomendado a partir de junio 2023! (sf)
icteri_file <- here("data","icteridae_maps","icteridae_maps.shp")
icteri_maps.sf <- st_read(icteri_file, stringsAsFactors = FALSE)Reading layer `icteridae_maps' from data source
`/home/luisd/Dropbox/darcyDB/PDRF/curso-R-biodiversidad/data/icteridae_maps/icteridae_maps.shp'
using driver `ESRI Shapefile'
Simple feature collection with 190 features and 26 fields
Geometry type: MULTIPOLYGON
Dimension: XY
Bounding box: xmin: -166.1927 ymin: -55.98218 xmax: -34.7901 ymax: 69.60669
Geodetic CRS: GCS_unknownCrear un vector con los nombres de todas las especies en el multipoligono
Creamos polígonos individuales por especie
Ahora sí, creamos el gradiente de riqueza con el paquete epm
icteriEPM <- createEPMgrid(spList, resolution = 1, retainSmallRanges = TRUE, extent = 'auto', method = 'percentOverlap', cellType = 'square', percentThreshold = 0.1)
|
| | 0%
|
|======================= | 33%
|
|=============================================== | 67%
|
|======================================================================| 100%¿Qué tiene el objeto generado?
Lo graficamos
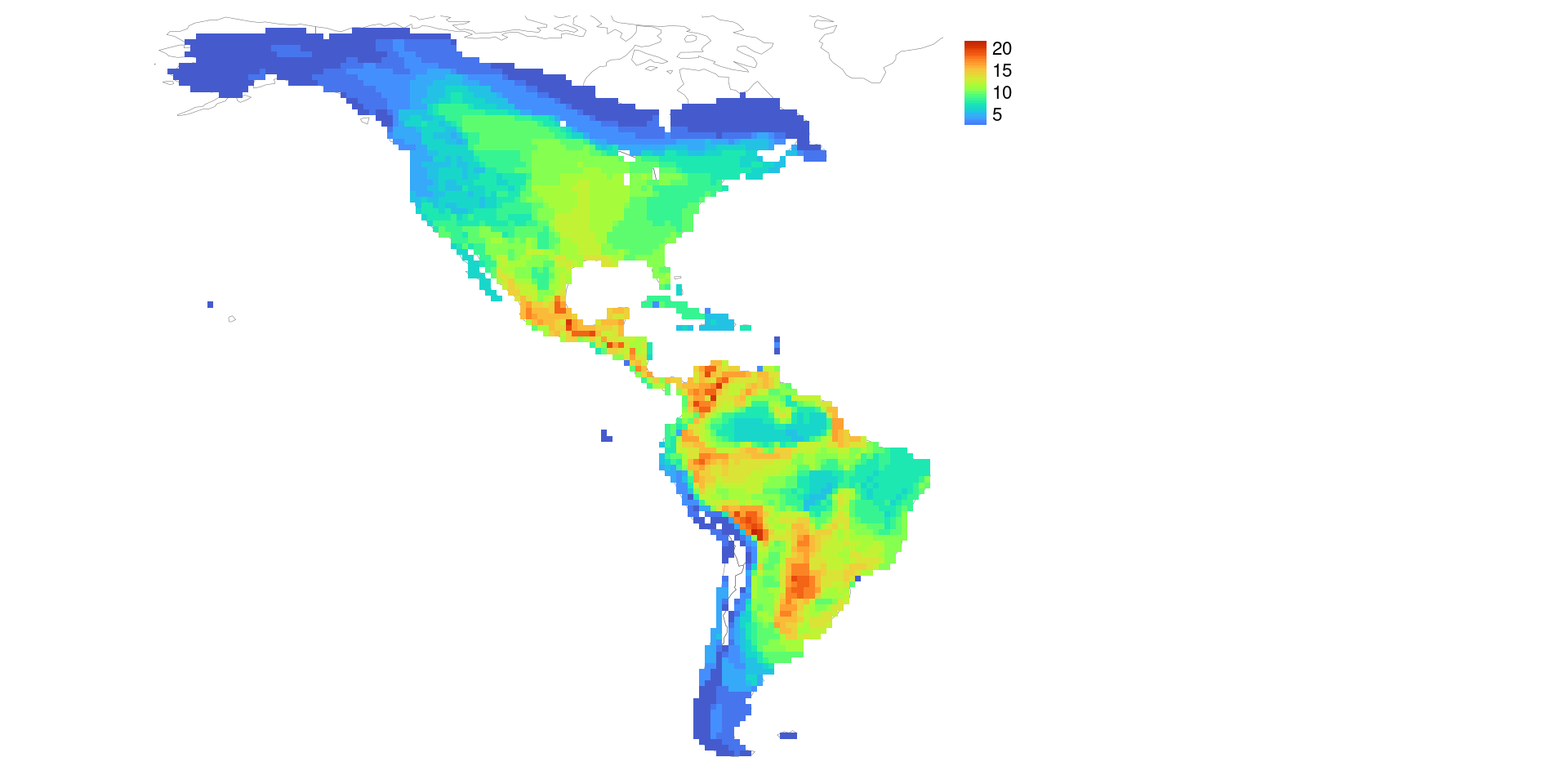
Ahora generamos la matriz de presencia-ausencia a partir del objeto creado. Esta matriz nos puede servir para comparar con los resultados de letsR o para derivar otras métricas/patrones